Desarrollar una escala predicitva de mortalidad (SIS) en pacientes con gripe grave considerando las variables de ingreso a UCI y comparar su eficacia respecto de un modelo d Random Forest (RF).
DiseñoSub-análisis de base de datos GETGAG/SEMICYUC.
ÁmbitoMedicina intensiva.
IntervencionesNinguna.
PacientesPacientes ingresados en 184 UCI españolas (2009-2018) con infección por gripe.
Variables: Demográficas, nivel de gravedad, tiempo síntomas hasta el ingreso al hospital (Gap-H) o desde hospital a UCI (Gap-UCI), o al diagnóstico (Gap-Dg), vacunación, cuadrantes infiltrados, insuficiencia renal, ventilación no-invasiva o invasiva (VM), shock y comorbilidades. Los puntos de corte y la importancia de las variables se obtuvieron de forma automática. Se realizó validación cruzada y regresión logística a partir de la cual se desarrolló la puntuación SIS. Se aplicó la puntuación y se calculó la exactitud y la discriminación (AUC-ROC) para SIS y RF. El análisis se realizó mediante CRAN-R Project.
ResultadosSe incluyeron 3.959 pacientes, edad 55 (43-67) años, 60% hombres, APACHE II de 16 (12-21) y SOFA 5 (4-8) puntos y una mortalidad del 21,3%. VM, shock, APACHE II, SOFA, insuficiencia renal aguda y Gap-UCI fueron incluidas en SIS. A partir de los OR se construyó el SIS que demostró una exactitud del 83% y un AUC-ROC del 82% similar al RF (AUC-ROC 82%).
ConclusionesLa escala SIS de fácil aplicación, ha demostrado una adecuada capacidad de estratificación del riesgo de mortalidad en la UCI. Sin embargo, estos resultados deberán ser validados prospectivamente.
To develop a mortality prediction score (Spanish Influenza Score [SIS]) for patients with severe influenza considering only variables at ICU admission, and compare its performance respect of Random Forest (RF).
DesignSub-analysis from the GETGAG/SEMICYUC database.
ScopeIntensive Care Medicine.
PatientsPatients admitted to 184 Spanish ICUs (2009-2018) with influenza infection Intervention: None.
VariablesDemographic data, severity of illness, times from symptoms onset until hospital admission (Gap-H), hospital to ICU (Gap-ICU) or hospital to diagnosis (Gap-Dg), antiviral vaccination, number of quadrants infiltrated, acute renal failure, invasive or noninvasive ventilation, shock and comorbidities. The study variable cut-off points and importance were obtained automatically. Logistic regression analysis with cross-validation was performed to develop the SIS score using the output coefficients. Accuracy and discrimination (AUC-ROC) were applied to evaluate SIS and RF. All analyses were performed using R (CRAN-R Project).
ResultsA total of 3959 patients were included. The mean age was 55 years (range 43-67), 60% were men, APACHE II 16 (12-21) and SOFA 5 (4-8), with ICU mortality 21.3%. Mechanical ventilation, shock, APACHE II, SOFA, acute renal failure and Gap-ICU were included in the SIS. The latter was generated according to the ORs obtained by logistic regression, and showed an accuracy of 83% with an AUC-ROC of 82%, similar to RF (AUC-ROC 82%).
ConclusionsThe SIS score is easy to apply and shows adequate capacity to stratify the risk of ICU mortality. However, further studies are needed to validate the tool prospectively.
La mortalidad de los pacientes críticos ingresado en UCI afectados por infección del virus de la gripe continúa siendo inaceptablemente elevada, algo superior al 20% en la población general1-3 y mayor al 30% en aquellos pacientes que requieren ventilación mecánica invasiva4. Las escalas utilizadas para predecir la gravedad en pacientes con neumonía comunitaria parecen subestimar la gravedad de los pacientes con infección por gripe5. La posibilidad de contar con predictores precoces de evolución, puede ser una herramienta de ayuda a la decisión clínica, al momento de la asistencia de estos pacientes críticos. Diferentes estudios han intentado descubrir predictores relacionados con la mortalidad de este colectivo particular de pacientes en UCI5-8. Sin embargo, la mayoría de ellos presentan graves limitaciones relacionadas con el pequeño número de pacientes incluidos6,7, la metodología empleada para obtener el predictor5,8 o aplicación a subpoblaciones especiales9,10. Desarrollar predictores de mortalidad en pacientes críticos es una tarea compleja, debido a su heterogeneidad y a la diferente respuesta sistémica ante la misma enfermedad. Las nuevas tecnologías informáticas permiten generar de forma automática, modelos de predicción mediante la utilización de estrategias de «machine learning».11-15. Sin embargo, la mayoría de estos modelos son poco comprensibles por los médicos, con una muy baja aceptación a las decisiones clínicas basadas en algoritmos crípticos (cajas negras) y en general, no tienen una clara aplicación en la práctica clínica16. Por ello, es importante desarrollar modelos predictivos que aprovechen estas nuevas tecnologías de análisis pero que sean comprensibles, precoces y de aplicación práctica, con la finalidad de ayudar en la decisión clínica. El objetivo de nuestro estudio es desarrollar mediante el uso de técnicas de machine learning, una escala gravedad, comprensible y aplicable (Spanish Influenza Score: SIS), que permita categorizar o estratificar el riesgo de mortalidad en forma precoz, en los pacientes con gripe al ingreso en la UCI.
Objetivo primarioDesarrollar mediante técnicas de machine learning, una escala de estratificación de la gravedad (SIS) y evaluar la capacidad de predicción de mortalidad en UCI para pacientes con gripe grave en UCI
Objetivo secundarioEvaluar la capacidad de predicción de la mortalidad de un modelo no lineal como Random Forest (RF) en pacientes con gripe grave en UCI respecto de la escala SIS.
Material y métodoTipo de estudioSubanálisis de base de datos GETGAG/SEMICYUC con pacientes ingresados en 184 UCI españolas en el periodo comprendido entre 2009-2018, con infección probada por gripe17.
Fuente de los datosEl conjunto de datos de entrenamiento (GE) y el de validación (GV) utilizados para el desarrollo del presente modelo, pertenecen a la base de datos creada en 2009 con motivo de la pandemia por la SEMICYUC para favorecer el conocimiento de esta nueva entidad y generar conocimientos de referencia para la mejor práctica clínica. El estudio fue aprobado por el Comité Ético de Investigación Clínica del Hospital Universitario Joan XXIII (IRB#11809) y ratificado por cada comité local de los centros participantes. La identidad de los pacientes permaneció anónima y no se solicitó consentimiento informado por la naturaleza observacional y epidemiológica del estudio, como ha sido publicado con anterioridad2,3,17-21.
Se incluyeron todos los pacientes ingresados en forma consecutiva en las 184 UCI Españolas participantes, con signos clínicos respiratorios sugestivos de infección viral, con o sin fiebre y con confirmación microbiológica de gripe A o B mediante PCR-rt2,3,17-21.
Los datos fueron obtenidos por los médicos asistenciales a través del examen físico, la revisión de la historia clínica, la radiología y los resultados de laboratorio. Los médicos asistenciales de cada centro fueron los responsables de solicitar todos los análisis y de realizar todos los procedimientos relacionados con el cuidado del paciente. Solamente se excluyeron del estudio pacientes menores de 15 años y aquellos pacientes con datos perdidos relacionados con los objetivos del estudio.
La base de datos contiene variables demográficas, nivel de gravedad, tiempo de inicio de los síntomas hasta el ingreso al hospital (Gap-H) o desde el ingreso al hospital hasta la UCI (Gap-UCI), o hasta el diagnóstico (Gap-Dg), vacunación antigripal, cuadrantes infiltrados en la radiografía de tórax, insuficiencia renal, ventilación no-invasiva (VNI) o invasiva (VM), fracaso a la VNI, shock al ingreso y comorbilidades, así como resultados laboratorio. La valoración de la severidad de la enfermedad se realizó a través del APACHE II score y el grado de disfunción orgánica a través del SOFA score.
Definiciones: las definiciones de las variables se pueden observar en la tabla 1 del anexo B y en publicaciones anteriores2,3,17-21.
Tratamiento de valores perdidosSe excluyeron los pacientes con datos perdidos en variables categóricas y se imputaron los valores perdidos de las variables numéricas mediante la función missForest/CRAN-R, una imputación no paramétrica del valor perdido utilizando árboles aleatorios22,23.
Elección de los puntos de corte de las variablesPara el análisis, se debió convertir las variables numéricas continuas en categóricas. Los puntos de corte para las variables numéricas se obtuvieron de forma automática a través de la función LOESS smoothing (paquete stats/CRAN R). La regresión de LOESS24 permite trazar curvas de una serie temporal mediante un método de regresión de mínimos cuadrados. Una vez obtenidas las curvas, se definen los puntos de corte a través de aquellas variaciones en la curva que se asocien con un aumento de al menos un 10% de la mortalidad.
Elección de las variables a incluir en el modeloLa elección de las variables se realizó de forma automática a través de la obtención del «information Value» para cada una de ellas mediante el paquete estadístico «InformationValue»-CRAN R. El Information Value (IV) es un método de búsqueda para seleccionar una variable predictora a través de regresión logística binaria25,26. El IV total es la suma de los IV de su categoría y una medida de la capacidad predictiva de una variable, para discriminar «casos o eventos» y los «controles o no eventos». Para el IV se consideró un punto de corte ≥ 0,20 para incluir las variables al modelo según lo sugeridos por Siddiqi26.
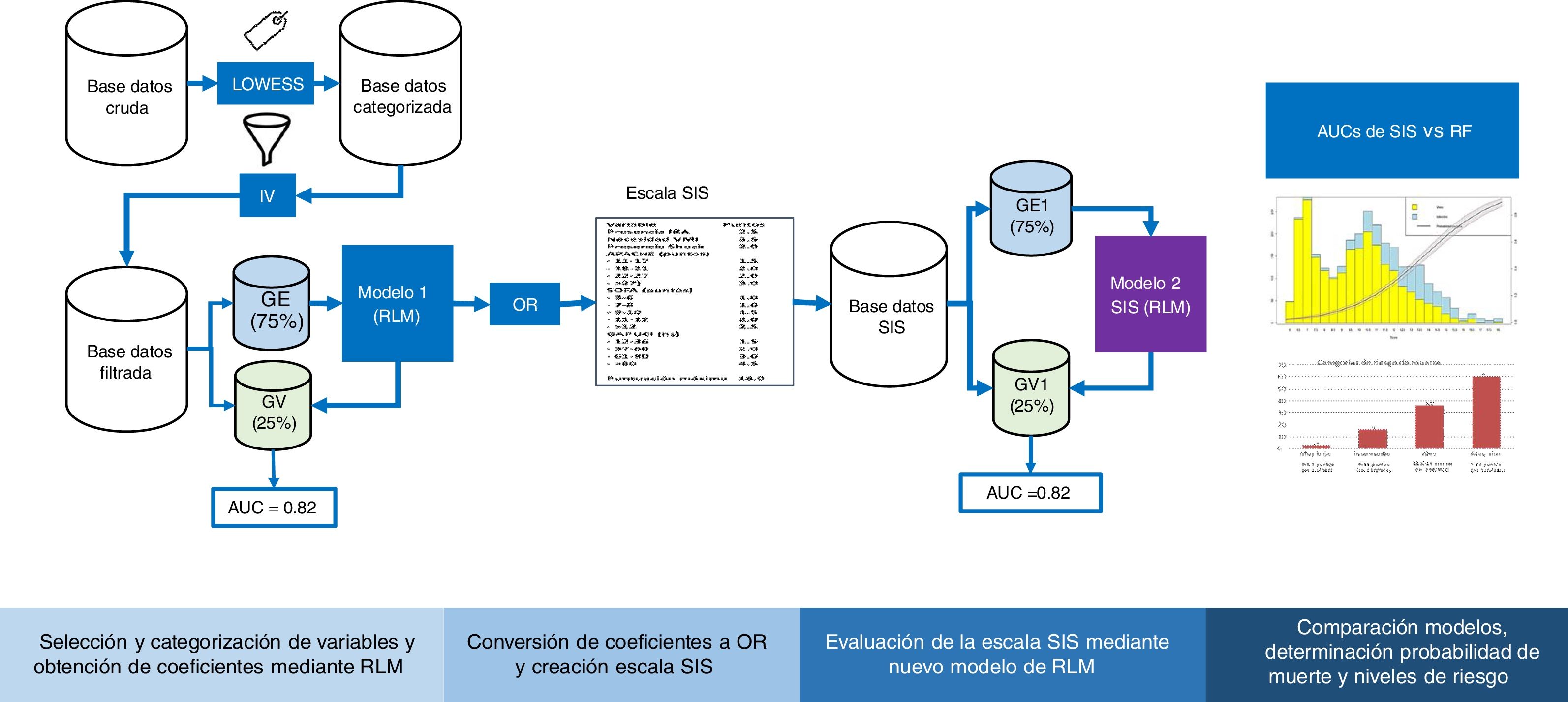
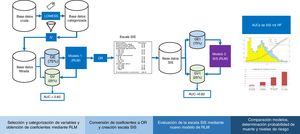
Validación cruzadaEn la figura 1 se puede ver el flujo del análisis del estudio. La cohorte original de pacientes se dividió en dos grupos, GE (75% de los pacientes) para crear el modelo y GV (25% restante) para evaluar la precisión y el error de dicho modelo. La partición se realizó de forma aleatoria, pero manteniendo una misma proporción en la variable respuesta «y» (mortalidad).
Diagrama de flujo del desarrollo y validación de la escala Spanish Influenza Score (SIS). LOWEES: Regresión de LOWEES; IV: information value; GE: grupo de entrenamiento; GV: grupo de validación; AUC: área bajo la curva ROC; RLM: Regresión logística múltiple; OR: Odds ratio; RF: Random Forest.
Una vez categorizadas todas las variables, se obtuvo un «valor» para cada nivel mediante un modelo binomial de regresión logística (RL) con la función «glm» de R. A partir de los coeficientes se obtuvieron los ODDS RATIO (OR), los cuales se redondearon para determinar los puntos asignados a cada variable del score SIS. Se aplicó la puntuación a cada uno de los pacientes y mediante la sumatoria, se obtuvo el puntaje final de la escala SIS. Se realizó dicho procedimiento para el GE y para GV y se evaluó la capacidad predictiva del modelo mediante su exactitud y discriminación mediante AUC ROC.
Conversión del puntaje en probabilidad de muerte y visualización de los resultadosPara obtener la probabilidad de muerte del puntaje SIS se estimaron mediante RL los coeficientes de la escala y la probabilidad del evento (mortalidad), utilizando los valores individuales de cada paciente. Posteriormente se construyó una figura de barras (Barplot) para representar los supervivientes y fallecidos según el puntaje obtenido por la escala SIS y también una curva de probabilidad del evento «mortalidad en UCI».
Validación de la escala SISEl adecuado funcionamiento de la escala SIS se evaluó mediante la exactitud y discriminación del modelo, así como sensibilidad (S), especificidad (E), valor predictivo positivo (VPP) y negativo (VPN). Además, se evaluó la calibración entre el riesgo predicho y el observado mediante el índice de Sommers27. Finalmente, se definieron cuatro categorías de riesgo estratificado según mortalidad.
Modelo no lineal de mortalidad Random Forest (RF)Mediante RF se determinó un modelo de mortalidad con las variables de ingreso a UCI. RF es una metodología muy usada dentro de la familia de algoritmos de aprendizaje automático o machine learning que se basa en la creación de múltiples árboles (bosques) de decisión, que se construyen mediante un algoritmo que introduce un modelo aleatorio de selección de las variables para reducir la correlación entre ellas28,29. La importancia de la variable se define como la influencia que tiene cada una de ellas al quitarla del modelo respecto de la predicción. El modelo final fue valorado mediante la exactitud, discriminación, S, E, VPP y VPN.
Expresión de los resultadosLos valores son expresados como medianas y rango intercuartil (RIC) 25-75%, o bien número y porcentajes según corresponda. Los resultados del análisis multivariado se expresan como Odd Ratio y su respectivo intervalo de confianza (IC) 95%. El análisis estadístico se realizó en el entorno del software R, versión 3.6.0 usando los paquetes de R.
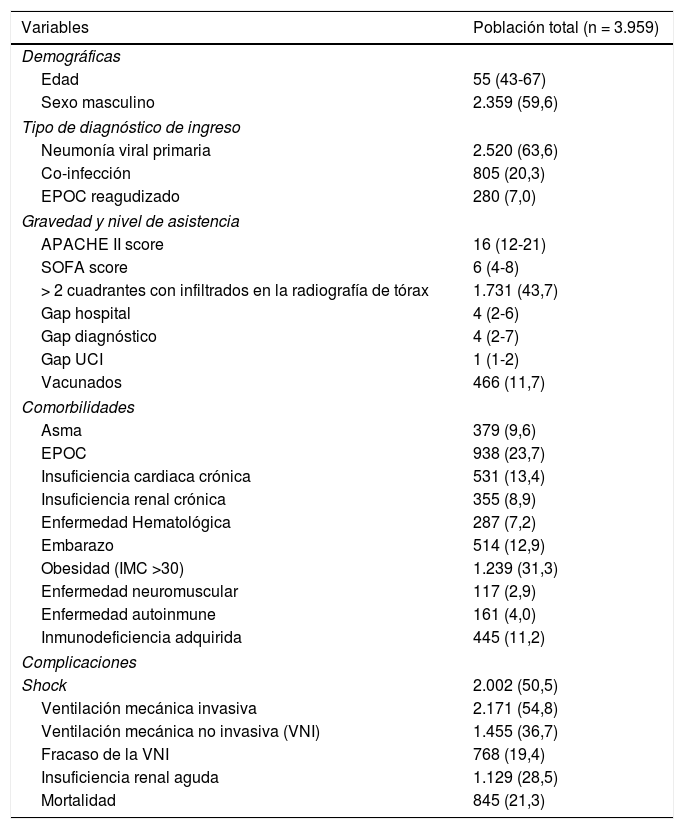
ResultadosPoblación generalLa cohorte total de estudio incluyó 3.959 pacientes ingresados en 184 UCI españolas. Las características generales de los pacientes se pueden ver en la tabla 1.
Características generales de los 3.959 pacientes incluidos en el presenta análisis. Las variables son aquellas consideradas al ingreso a UCI hasta las primeras 24 horas de evolución (los resultados están expresado en número de pacientes (n) y porcentaje (%) o mediana y rango intercuartil (RIC) según corresponda. EPOC: enfermedad pulmonar obstructiva crónica; APACHE II: Acute Physiology And Chronic Health Evaluation; SOFA: Sequential Organ Failure Assessment; Gap hospital: tiempo desde inicio de los síntomas hasta ingreso en el Hospital; Gap diagnóstico: tiempo desde ingreso al Hospital y el diagnóstico; Gap UCI: tiempo desde el ingreso al hospital hasta la Unidad de Cuidados Intensivos-UCI; vacunados: pacientes que recibieron vacunación antigripal; IMC: índice de masa corporal)
| Variables | Población total (n = 3.959) |
|---|---|
| Demográficas | |
| Edad | 55 (43-67) |
| Sexo masculino | 2.359 (59,6) |
| Tipo de diagnóstico de ingreso | |
| Neumonía viral primaria | 2.520 (63,6) |
| Co-infección | 805 (20,3) |
| EPOC reagudizado | 280 (7,0) |
| Gravedad y nivel de asistencia | |
| APACHE II score | 16 (12-21) |
| SOFA score | 6 (4-8) |
| > 2 cuadrantes con infiltrados en la radiografía de tórax | 1.731 (43,7) |
| Gap hospital | 4 (2-6) |
| Gap diagnóstico | 4 (2-7) |
| Gap UCI | 1 (1-2) |
| Vacunados | 466 (11,7) |
| Comorbilidades | |
| Asma | 379 (9,6) |
| EPOC | 938 (23,7) |
| Insuficiencia cardiaca crónica | 531 (13,4) |
| Insuficiencia renal crónica | 355 (8,9) |
| Enfermedad Hematológica | 287 (7,2) |
| Embarazo | 514 (12,9) |
| Obesidad (IMC >30) | 1.239 (31,3) |
| Enfermedad neuromuscular | 117 (2,9) |
| Enfermedad autoinmune | 161 (4,0) |
| Inmunodeficiencia adquirida | 445 (11,2) |
| Complicaciones | |
| Shock | 2.002 (50,5) |
| Ventilación mecánica invasiva | 2.171 (54,8) |
| Ventilación mecánica no invasiva (VNI) | 1.455 (36,7) |
| Fracaso de la VNI | 768 (19,4) |
| Insuficiencia renal aguda | 1.129 (28,5) |
| Mortalidad | 845 (21,3) |
A través de la Regresión de LOESS se dibujaron las curvas para las variables continuas como el APACHE II, SOFA score y Gap-UCI (fig. 1 en anexo B). En base al cambio del 10% en la probabilidad de muerte en cada curva, se establecieron los siguientes puntos de corte: a) para el APACHE II, 4 puntos de corte: 1) 11 a 17, 2) 18 a 21, 3) 22 a 27 y 4) más de 27 puntos; b) para el SOFA, 5 puntos de corte: 1) 3 a 6, 2) 7 a 8, 3) 9 a 10, 4) 11 a 12 y 5) mayor de 12 puntos y c) para el Gap-UCI, los días se transformaron en horas y se determinaron 4 puntos de corte: 1) 12-36, 2) 37-60, 3) 61-80 y 4) más de 80 horas. Estos puntos de corte se incluyeron en el modelo de regresión.
Elección de las variables mediante el valor de la información (information value = IV)La capacidad de predicción de cada variable respecto de la mortalidad en UCI fue evaluada mediante método de regresión lineal para obtener el IV. Solamente ventilación mecánica invasiva (VMI), SOFA score, APACHE II score, shock, insuficiencia renal aguda (IRA) y Gap-UCI fueron las variables que llegaron al punto de corte establecido para ser incluidas en el modelo. (en anexo B).
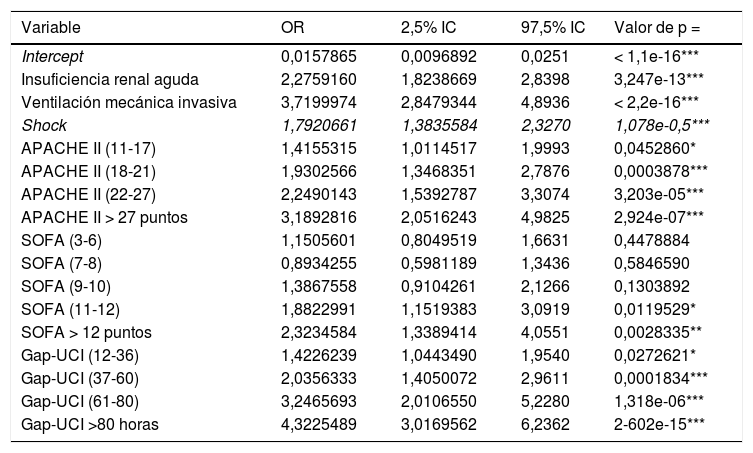
Modelo de regresiónSe dividió a la población en un grupo de entrenamiento (GE; n = 2.970) y en un grupo de validación (GV; n = 989). Las características de cada grupo pueden observase en la tabla 3 del anexo B. Los puntos de corte establecidos para APACHE II, SOFA y Gap-UCI y las variables categóricas ventilación VMI, shock e IRA se incluyeron en el modelo de regresión. En la tabla 2 se pueden observar las variables asociadas de forma independiente con la mortalidad. Una vez aplicado el modelo en el GV, se observó una exactitud del 82% y un AUC ROC 82%.
Variables asociadas de forma independiente con la mortalidad en UCI (análisis multivariado) (APACHE II: Acute Physiology And Chronic Health Evaluation; SOFA: Sequential Organ Failure Assessment; Gap UCI: tiempo desde el ingreso al hospital hasta la Unidad de Cuidados Intensivos)
| Variable | OR | 2,5% IC | 97,5% IC | Valor de p = |
|---|---|---|---|---|
| Intercept | 0,0157865 | 0,0096892 | 0,0251 | < 1,1e-16*** |
| Insuficiencia renal aguda | 2,2759160 | 1,8238669 | 2,8398 | 3,247e-13*** |
| Ventilación mecánica invasiva | 3,7199974 | 2,8479344 | 4,8936 | < 2,2e-16*** |
| Shock | 1,7920661 | 1,3835584 | 2,3270 | 1,078e-0,5*** |
| APACHE II (11-17) | 1,4155315 | 1,0114517 | 1,9993 | 0,0452860* |
| APACHE II (18-21) | 1,9302566 | 1,3468351 | 2,7876 | 0,0003878*** |
| APACHE II (22-27) | 2,2490143 | 1,5392787 | 3,3074 | 3,203e-05*** |
| APACHE II > 27 puntos | 3,1892816 | 2,0516243 | 4,9825 | 2,924e-07*** |
| SOFA (3-6) | 1,1505601 | 0,8049519 | 1,6631 | 0,4478884 |
| SOFA (7-8) | 0,8934255 | 0,5981189 | 1,3436 | 0,5846590 |
| SOFA (9-10) | 1,3867558 | 0,9104261 | 2,1266 | 0,1303892 |
| SOFA (11-12) | 1,8822991 | 1,1519383 | 3,0919 | 0,0119529* |
| SOFA > 12 puntos | 2,3234584 | 1,3389414 | 4,0551 | 0,0028335** |
| Gap-UCI (12-36) | 1,4226239 | 1,0443490 | 1,9540 | 0,0272621* |
| Gap-UCI (37-60) | 2,0356333 | 1,4050072 | 2,9611 | 0,0001834*** |
| Gap-UCI (61-80) | 3,2465693 | 2,0106550 | 5,2280 | 1,318e-06*** |
| Gap-UCI >80 horas | 4,3225489 | 3,0169562 | 6,2362 | 2-602e-15*** |
Significación *** 0.001; ** 0.01: *0.05
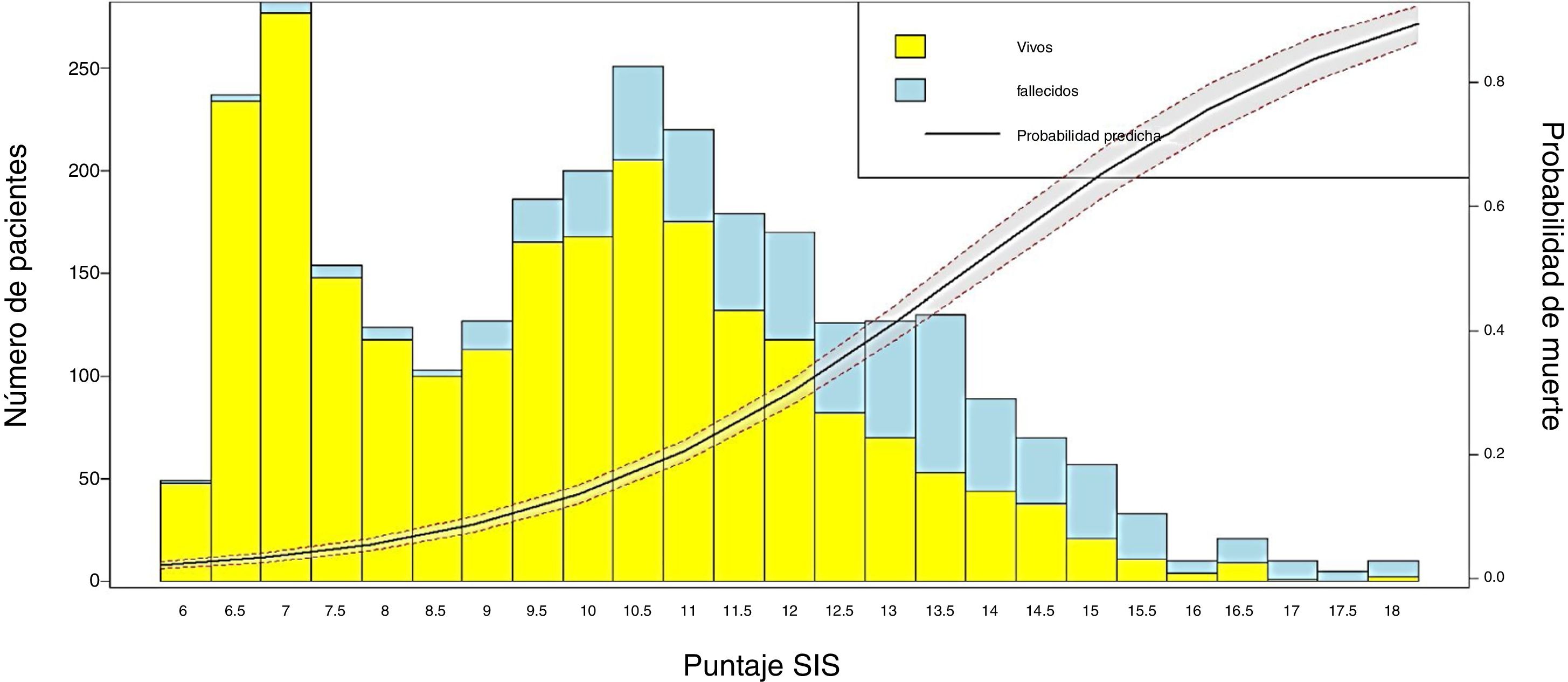
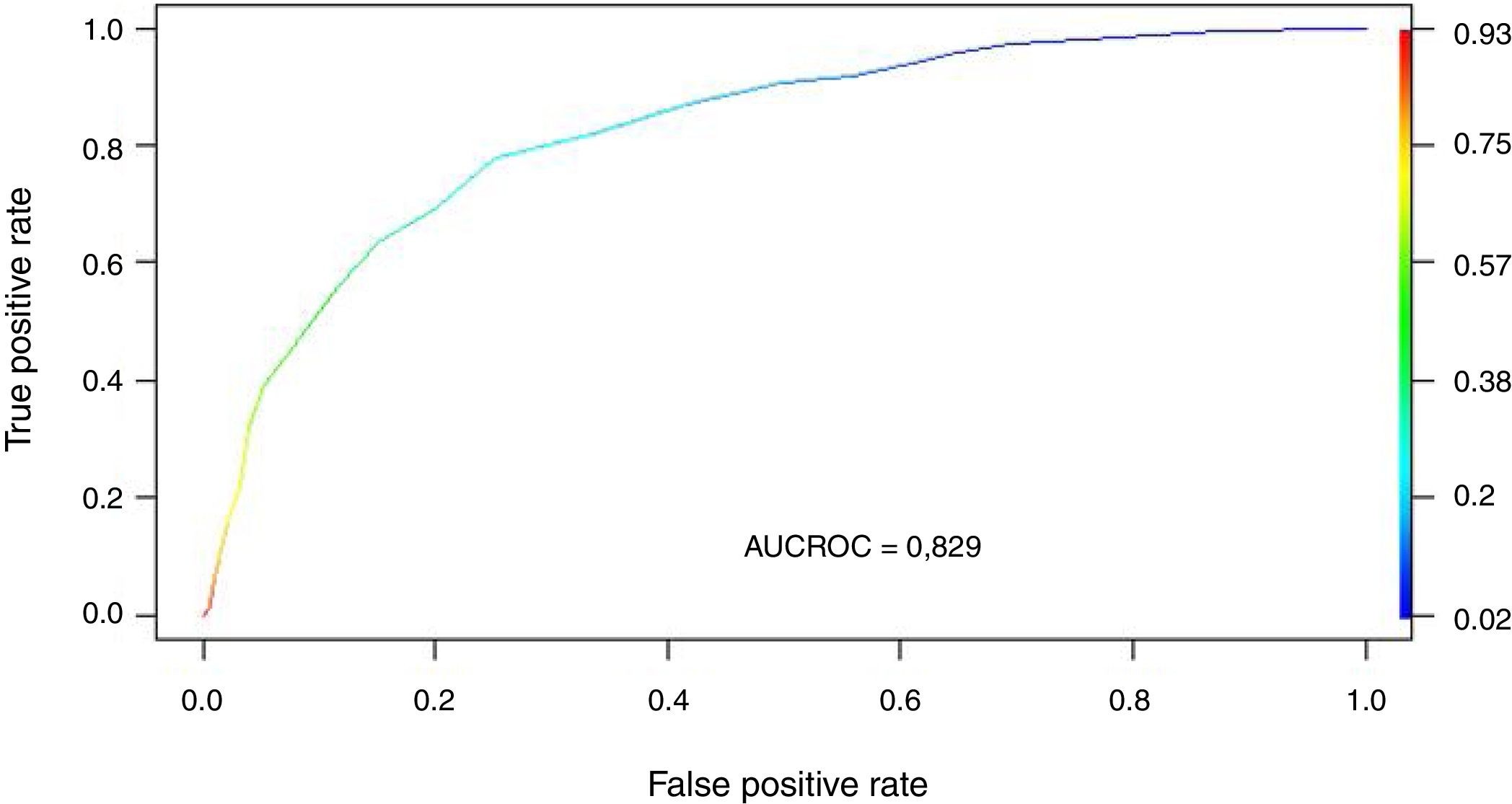
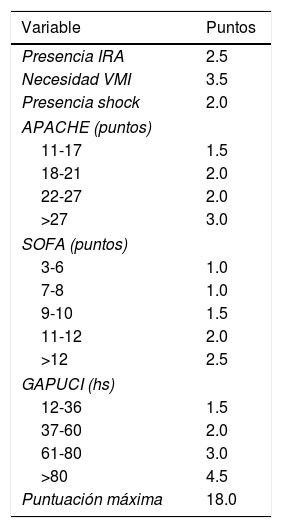
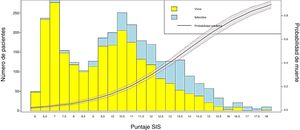
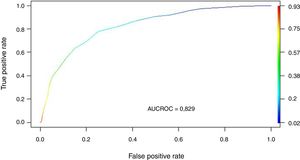
Se transformaron los OR de cada variable en los puntos del score mediante redondeo al 0,5 más cercano y se desarrolló un score con un puntaje máximo de 18 puntos (tabla 3). Se aplicó el puntaje a cada uno de los pacientes del GD y se obtuvo la predicción de mortalidad respecto de la puntuación para cada paciente (fig. 2). Después se aplicó el score al GV y se obtuvo una exactitud del 83% (95% IC: 0,79-0,84) con un área bajo la curva ROC del 82% (fig. 3) demostrando una buena discriminación del SIS. En la figura 2 del anexo B se puede observar que la calibración del modelo es buena con un índice de Sommers de 0,65 y en la tabla 4 se observan los valores predictivos del SIS.
Puntaje del Spanish Influenza Score (SIS) derivado de los OR de la regresión logística (IRA: Insuficiencia renal aguda; VMI: ventilación mecánica invasiva; APACHE II: Acute Physiology And Chronic Health Evaluation; SOFA: Sequential Organ Failure Assessment;Gap UCI: tiempo desde el ingreso al hospital hasta la Unidad de Cuidados Intensivos)
| Variable | Puntos |
|---|---|
| Presencia IRA | 2.5 |
| Necesidad VMI | 3.5 |
| Presencia shock | 2.0 |
| APACHE (puntos) | |
| 11-17 | 1.5 |
| 18-21 | 2.0 |
| 22-27 | 2.0 |
| >27 | 3.0 |
| SOFA (puntos) | |
| 3-6 | 1.0 |
| 7-8 | 1.0 |
| 9-10 | 1.5 |
| 11-12 | 2.0 |
| >12 | 2.5 |
| GAPUCI (hs) | |
| 12-36 | 1.5 |
| 37-60 | 2.0 |
| 61-80 | 3.0 |
| >80 | 4.5 |
| Puntuación máxima | 18.0 |
Gráfico de barras que muestra la aplicación de la puntuación obtenida SIS y la mortalidad observada, así como la curva de probabilidad de mortalidad para cada nivel. Se observa que la mortalidad se incrementa significativamente a medida que lo hace la puntuación obtenida (p < 0,001).
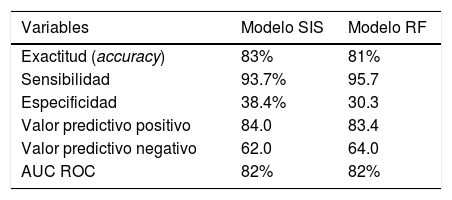
Valores predictivos del Spanish Influenza Score (SIS) y del modelo mediante Random Forest (RF) para los 3.959 pacientes incluidos
| Variables | Modelo SIS | Modelo RF |
|---|---|---|
| Exactitud (accuracy) | 83% | 81% |
| Sensibilidad | 93.7% | 95.7 |
| Especificidad | 38.4% | 30.3 |
| Valor predictivo positivo | 84.0 | 83.4 |
| Valor predictivo negativo | 62.0 | 64.0 |
| AUC ROC | 82% | 82% |
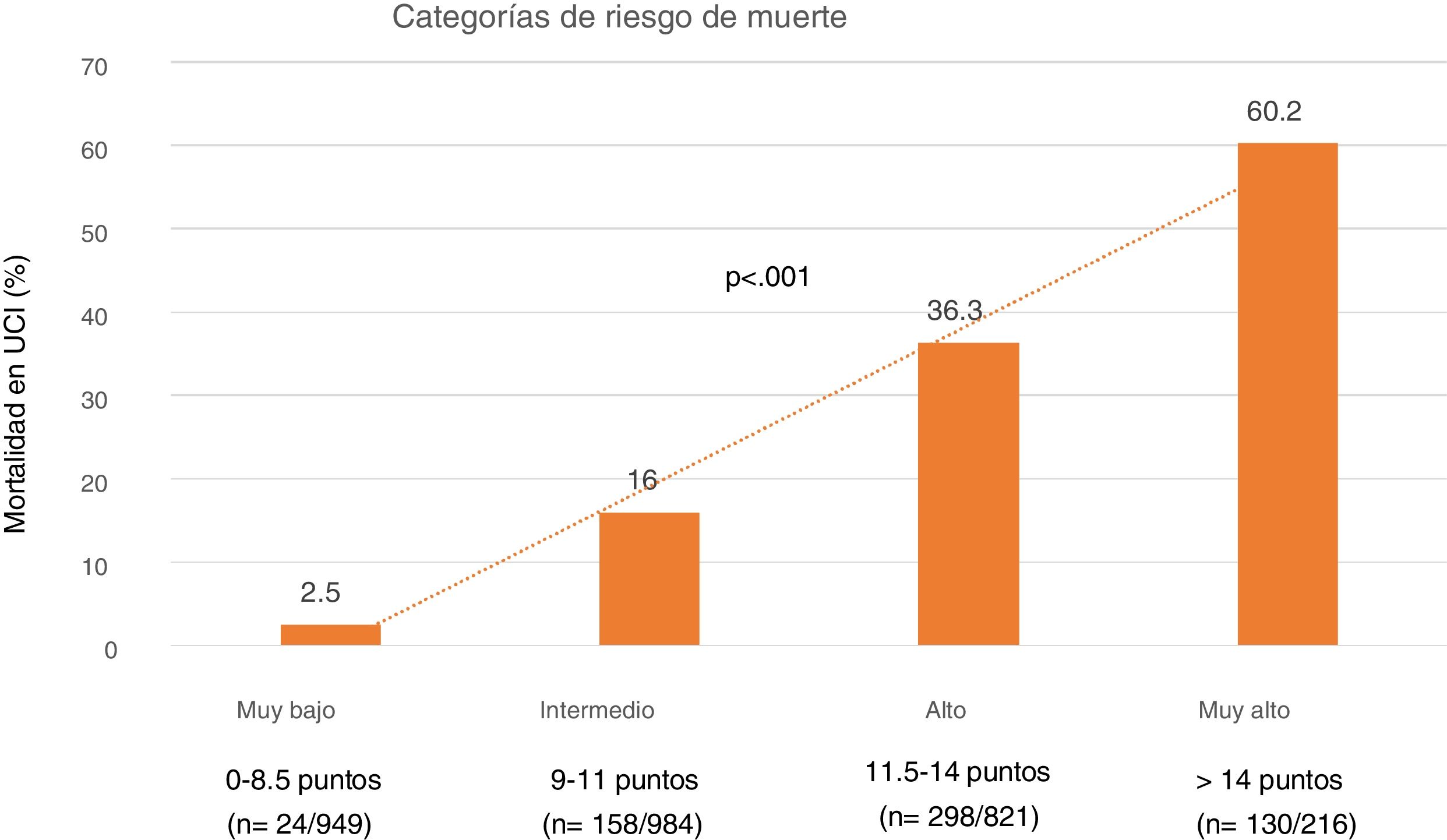
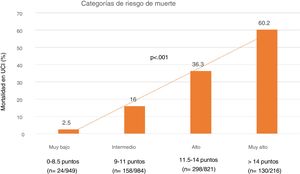
Finalmente, se determinaron cuatro niveles de riesgo de SIS estratificados según mortalidad: 1) Muy bajo riesgo: SIS 0-8.5 puntos con una mortalidad del 2,5%; 2) Riesgo moderado: SIS 9-11 puntos con una mortalidad del 16%; 3) Riesgo alto: SIS 11,5 -14 puntos con una mortalidad del 36.3% y 4) Muy alto riesgo: SIS > 14 puntos con una mortalidad del 60% (fig. 4).
Modelo de predicción de mortalidad mediante Random Forest (modelo no lineal)La aplicación de RF evidenció a la ventilación mecánica invasiva, SOFA score, insuficiencia renal aguda, días hasta la UCI, APACHE II, fracaso de la ventilación no invasiva e inmunodeficiencia como las variables con mayor impacto sobre la predicción (fig. 3 del anexo B). La valoración de la predicción evidenció una exactitud del 81% con un AUC ROC de 82%. En la tabla 4 se muestran el resto de los parámetros de predicción y su comparación respecto del SIS.
DiscusiónLa aplicación de escalas de gravedad a nivel individual y poblacional es de vital importancia pues permite clasificar y estratificar a los pacientes en categorías de riesgo, en función de uno de los resultados más importantes que pueden medirse en la UCI como es la mortalidad. Sobre este concepto, el objetivo principal de nuestro estudio fue desarrollar un modelo de predicción «precoz» de mortalidad, mediante aplicación de métodos de machine learning y comparar su rendimiento respecto de un modelo no lineal de Random Forest.
El principal hallazgo del estudio fue que la escala SIS desarrollada, demostró una adecuada exactitud en la validación cruzada (83%) y una muy buena discriminación (AUCROC 82%), parámetros de predicción similares al modelo Random Forest. Estos datos sugieren que la escala SIS es un modelo válido, que permite estratificar adecuadamente el riesgo de muerte en pacientes con gripe al ingreso a UCI.
Hasta el momento los estudios realizados solo han determinado variables asociadas a la mortalidad a través de análisis multivariado clásico5,6,19,30 o bien, desarrollando escalas con un limitado número de pacientes7,31-33 o considerando solamente poblaciones especiales9,10. Oh et al.7 en 709 pacientes, desarrollaron una escala con 4 variables, adjudicando un punto para cada una de ellas (estado mental alterado, hipoxia, infiltrados bilaterales y edad > 65 años). Aunque es un estudio multicéntrico y la discriminación de la escala fue muy buena (AUC ROC 0,83), solo 75 (10,5%) pacientes fueron casos graves. Además, los autores no realizaron validación cruzada. Adeniji et al.8 aplicaron el STSS (Simple Triage Scoring System)31 y el SOFA en urgencias, para predecir la necesidad de ventilación mecánica (VM) y el ingreso en UCI de pacientes con gripe. La discriminación fue superior para el STSS (AUC ROC 0,88) respecto del SOFA (AUC ROC 0,77) para el ingreso a UCI y también para necesidad de VM (AUC ROC 0,91 vs. AUC ROC 0,87 para STSS y SOFA respectivamente). Sin embargo, el número de pacientes incluidos es muy bajo (n = 62), por lo cual la potencia del estudio es pobre y los resultados difíciles de interpretar. Chung et al.9 desarrollaron una escala de gravedad en 409 pacientes ancianos (GID: Geriatric Influenza Death). El análisis multivariado evidenció solo cinco variables (coma, elevación de la PCR, cáncer, enfermedad coronaria y presencia de bandas en la fórmula leucocitaria) asociadas de forma independiente con la mortalidad. Aunque el GID, mostró una muy buena discriminación (AUC ROC 86%), a diferencia de nuestra escala, considera variables de toda la evolución, se limita a la población de mayores y no se ha realizado validación cruzada.
Los estudios basados en métodos estadísticos habituales, como la regresión logística (modelo lineal), son ampliamente aceptados por los médicos para determinar o investigar factores relacionados con la mortalidad o el desarrollo de algún evento adverso. Sin embargo, estos indicadores no funcionan adecuadamente para la predicción individual15, ni permiten predecir el rumbo clínico de un paciente. Nuevas formas de predicción, utilizando algoritmos desarrollados mediante técnicas de aprendizaje automático (machine learning), como las redes neuronales o los árboles de decisión, han sido implementados para la obtención de modelos de predicción en diferentes escenarios en cuidados intensivos34-37. Pero estos modelos, aunque demuestran muy buen rendimiento en la predicción, suelen ser incomprensibles para los clínicos y poco aplicables, no solo por su complejidad, sino también, por la falta de inclusión en el modelo de variables de gran interés clínico, como el tratamiento antimicrobiano, en un modelo complejo que compara modelos clínicos versus automáticos en el tratamiento de la sepsis38, lo cual invalida la aplicación clínica del modelo. De forma reciente, Hu et al.39 publicaron un estudio sobre la aplicación de dos técnicas de machine learning (gradient boosting XGBoost y RF) comparadas con un modelo de RL para predecir la mortalidad a los 30 días, en una cohorte de 336 pacientes con gripe. Los autores concluyen que los modelos XGBoost (AUC ROC = 0,84) y RF(AUC ROC = 0,80) presentaron una mejor discriminación respecto de la RL (AUC ROC = 0,70). Estos resultados no se corresponden con lo observado en nuestro estudio, donde también utilizamos un modelo de RF. La discrepancia podría estar relacionada con el bajo número de pacientes respecto de un elevado número de variables consideradas en el estudio de Hu et al.39, lo cual afecta de forma desfavorable a los modelos de regresión, pero no a los desarrollados mediante arboles de decisión. Además, utiliza variables de los primeros 7 días, con lo cual no puede considerarse como un predictor precoz. Finalmente, la discriminación del mejor modelo (XGBoost), el cual es poco interpretable para el clínico, solo es ligeramente superior a la observada en la escala SIS.
Las escalas utilizadas de forma habitual en UCI para medir la gravedad general (APACHE II) o el grado de disfunción orgánica (SOFA) presentan limitaciones al momento de categorizar los pacientes con gripe grave7,8,39-41, por lo cual nuestra escala SIS, podría ser una alterativa sencilla a implementar en este grupo de pacientes, ya que ha demostrado un rendimiento similar a un modelo de predicción desarrollado con Random Forest (RF). Aunque el RF es uno de los mejores métodos de machine learning para responder a los problemas complejos, especialmente aquellos relacionados con asociaciones no lineales29, la principal desventaja del método es que es poco entendible para el clínico, ya que no permite conocer cómo se realizan las asociaciones (cajas negras) para generar el modelo final. En el mismo sentido que nuestros resultados, pero en la población general de UCI, Kim et al.15 estudiaron la capacidad de predicción de mortalidad, de tres diferentes modelos desarrollados con machine learning (redes neuronales, support vector machine y árboles de decisión), respecto de un modelo tradicional de regresión logística desarrollado con las variables del APACHE III score42. El estudio incluyó a más de 38.000 ingresos y solo consideró los datos capturados en las primeras 24 horas de ingreso a UCI. El estudio evidenció una capacidad de predicción similar entre los cuatro modelos, resaltando a la regresión logística como un método válido para determinar la predicción de mortalidad respecto de modelos más complejos.
Nuestro estudio combina técnicas de aprendizaje automático con regresión logística, lo cual le brinda robustez y objetividad. Además, siendo un estudio multicéntrico y con un elevado número de pacientes, permite la generalización de los resultados, ya que las 184 UCI participantes representan aproximadamente el 50% de todas las UCI de España. Sin embargo, nuestro estudio presenta algunas limitaciones que deben ser remarcadas para su adecuada interpretación. Primero, nuestra escala SIS utiliza solamente datos al ingreso de UCI, por lo cual, datos relacionados con los cambios producidos durante la evolución no son considerados. Aunque esto puede afectar la capacidad de predicción, nuestro objetivo primario fue desarrollar una escala de riesgo «precoz» al momento de ingreso a UCI y no durante su evolución, la cual ha demostrado un adecuado poder de discriminación. Segundo, el desarrollo del modelo se ha realizado considerando solamente pacientes ingresados en UCI españolas, por lo cual, el modelo podría no presentar un adecuado funcionamiento en otros países, o en otras poblaciones fuera del ámbito de la UCI. Tercero, aunque se ha realizado una validación cruzada, el funcionamiento de SIS no ha sido probado en forma prospectiva, por lo cual, nuestro proyecto contempla una validación prospectiva nacional e internacional del SIS para evaluar el impacto clínico real y la aceptación de la escala por parte de los intensivistas.
En conclusión, la escala SIS desarrollada en base a los datos de más de 3.900 pacientes críticos, demuestra una predicción similar a la observada para un modelo de Random Forest. Considerando que la aplicabilidad de la escala SIS es sencilla y permite obtener una estratificación del riesgo de muerte de forma precoz, su implementación podría impactar favorablemente en la evolución de los pacientes ingresados en UCI por infección grave por gripe. Sin embargo, estas consideraciones deberán ser confirmadas con la validación prospectiva del SIS.
Autoría/colaboracionesConcepción y el diseño del estudio: AR (Alejandro Rodriguez), ED (Emili Díaz) ST(Sandra Trefler), JMC (Judth Marín-Corral), LC(Laura Claverias). IML(Ignacio Martín Loeches),MB(María Bodi), JSV(Jordi Sole-Violan), JG(Jose garnacho-Montero), MRB (Manuel Ruiz Botella), JG (Josep Gomez), JA(Jordi Albiol), EM (Eduard mallol)
Adquisición de los datos y análisis: AR (Alejandro Rodriguez), ST(Sandra Trefler), LC(Laura Claverias), GM(Gerard Moreno), MS (Manuel Samper), MB(María Bodi), JMC (Judth Marín-Corral), MRB (Manuel Ruiz Botella), JG (Josep Gomez), JA(Jordi Albiol), EM (Eduard mallol) AB(Ariel Barrios)
Interpretación de los datos: AR (Alejandro Rodriguez), MS (Manuel Samper), GM(Gerard Moreno), MB(María Bodi), ED (Emili Díaz) ST(Sandra Trefler), JMC (Judth Marín-Corral), LC(Laura Claverias), JCY (Juan carlos yebenes), AT (Antoni Torres) PR(Paula Ramirez) JGM(Jose Garnacho Montero), RF(Ricard ferrer), IML(Ignacio Martín Loeches, LFR(Luis Felipe Reyes) JG(JUan Guardiola), MIR(Marcos I restrepo), JSV(Jordi Sole-Violan)
Importante aportación intelectual al contenido: AT (Antoni Torres), JGM (Jose Garnacho Montero), PR (Paula Ramirez), JCY (Juan Carlos Yebenes), RF (Ricard ferrer), LFR(Luis Felipe Reyes), JG (JUan Guardiola),MIR(Marcos I Restrepo)
Borrador del manuscrito: AR (Alejandro Rodriguez), MS (Manuel Samper), GM(Gerard Moreno)
Revisión crítica del contenido: MB(María Bodi), ED (Emili Díaz). JMC (Judth Marín-Corral), LC(Laura Claverias), JCY (Juan carlos yebenes), JSV(Jordi Sole-Violan), AT (Antoni Torres), PR (Paula Ramirez), JGM (Jose Garnacho Montero), RF (Ricard ferrer), IML(Ignacio Martín Loeches), LFR(Luis Felipe Reyes), AB(Ariel Barrios), JG(JUan Guardiola), MIR (Marcos I restrepo)
Todos los autores aprobaron el manuscrito final enviado para su evaluación y posible publicación.
Los hallazgos y las conclusiones del presente manuscrito son responsabilidad de los autores y no representan necesariamente la posición oficial de la SEMICYUC.
Papel de los auspiciantesLa SEMICYUC no ha tenido ninguna implicación en el diseño del estudio, en el análisis y la interpretación de los datos o en la redacción del presente manuscrito. JG ha tenido un tiempo parcial protegido destinado al análisis del estudio, a través de una beca de investigación de la Fundación Privada Barri. AR, el autor para la correspondencia, ha tenido acceso a todos los datos del estudio y es el responsable final de enviar el manuscrito para su publicación.
Conflicto de interesesAR ha recibido una beca de investigación de Gilead Science para el estudio de antibióticos nebulizados. Además, ha recibido honorarios por conferencias docentes para Biomerieux, Astellas, Pfizer, Thermo Fisher, MSD, Gilead, Shionogi y BRHAMS. Sin embargo, no tiene conflictos de interés con el presente manuscrito
Este estudio está auspiciado por la SEMICYUC (Sociedad Española de Medicina Intensiva, Crítica y Unidades Coronarias. Los autores agradecen a todos los investigadores del GETGAG (Grupo Español de Trabajo Gripe A Grave) por su continua participación en el proyecto desde el año 2009, sin la cual este proyecto no podría haberse realizado.
Coordinadores clínicos: M. Samper, G. Moreno, M. Bodi, E. Díaz, J. Marín-Corral, L. Claverias, S. Trefler, J.C. Yebenes, J. Solé Violán, A. Torres, P. Ramírez, J. Garnacho-Montero, R. Ferrer, A. Rodríguez
Coordinadores científicos de datos: M. Ruíz-Botella, J. Gómez, J. Albiol, E. Mayol
Consultores externos: I. Martín-Loeches, L.F. Reyes, A. Barrios, J. Guardiola, M.I. Restrepo