Editado por: F. Baigorri-González y J.º. Lorente Balanza
Última actualización: Abril 2005
Más datosINTRODUCCION
La sepsis constituye en la actualidad la principal causa de muerte en los pacientes ingresados en las Unidades de Cuidados Intensivos y su incidencia ha aumentado en los últimos años. A pesar de los potentes antimicrobianos que disponemos, de los avances en las técnicas de soporte hemodinámico, renal y respiratorio así como de algunos recientes tratamientos para modular la respuesta inflamatoria como es la proteína C activada, la mortalidad de la sepsis grave y el shock séptico continúan siendo muy elevadas. Si bien esta mortalidad puede reducirse con una resucitación precoz y con un tratamiento antibiótico adecuado, muchos pacientes correctamente tratados fallecen1. En algunos de ellos, las enfermedades previas pueden explicar la fatal evolución. No obstante, en muchos casos no tenemos una explicación clara de por qué algunos pacientes que sufren una sepsis grave evolucionan mal o por qué algunos enfermos con sepsis progresan a sepsis grave y shock séptico mientras otros con similar terapéutica no lo hacen.
En los últimos años y con los avances en el conocimiento del genoma humano se ha prestado un gran interés en la susceptibilidad genética a las infecciones. Uno de los hallazgos más destacados de nuestro genoma es la elevadísima frecuencia de secuencias de ADN no codificadoras. Sólo el 1,1 a 1,4 de nuestro genoma humano de 3,2 gigabase se transcribe a ARN mensajero para codificar una proteína. Otro de los hallazgos más trascendentes es la elevada variabilidad en el genoma humano. Una de las principales causas de esta variabilidad es lo que conocemos como polimorfismo genético. Algunos de estos cambios se traducen en una modificación de la secuencia de aminoácidos de la proteína que codifica y en cambios en la producción de dicha proteína.
En el presente trabajo nos proponemos 4 objetivos:
1. Revisar la nomenclatura y los conceptos básicos de la genética relacionados con el polimorfismo genético.
2. Enumerar los polimorfismos más frecuentes descritos en los mediadores implicados en la respuesta a la agresión.
3. Analizar los trabajos que han estudiado la relación entre los diversos polimorfismos y la mortalidad en pacientes con sepsis.
4. Evaluar los estudios que se han diseñado para conocer si determinados polimorfismos se asocian a un mayor riesgo de desarrollo de sepsis tras una agresión como puede ser la gran cirugía o el traumatismo grave.
POLIMORFISMO GENÉTICO: CONCEPTOS
Un polimorfismo genético (PG) es una variante alélica que existe de forma estable en una población. Para ser considerado un PG, debe presentar una frecuencia de al menos el 1%. Son, por lo tanto, diferentes de las mutaciones, que son mucho menos frecuentes y van asociadas, habitualmente, a enfermedades hereditarias. Se pueden distinguir dos tipos de PG: los polimorfismos de repetición en tándem (VNTR, del inglés variable number of tandem repeats) y los polimorfismos de un solo nucleótido (SNP, del inglés single nucleotide polymorphisms).
Existen dos tipos de polimorfismos de repetición, los VNTR-minisatélites y los VNTR-microsatélites. Los minisatélites son loci que corresponden a secuencias de ADN de unas pocas decenas de nucleótidos repetidas en tándem. El número de dichas repeticiones varía de cromosoma a cromosoma, por lo que cada loci puede presentar muchos alelos distintos (tantos como repeticiones). Los minisatélites presentan el inconveniente de no estar distribuidos por todo el genoma y por lo tanto sólo pueden ser utilizados en el diagnóstico de un número muy reducido de enfermedades. Los microsatélites corresponden a la repetición en tándem de secuencias de entre 2 y 5 nucleótidos. Los microsatélites presentan dos características que los hacen ideales para su uso. En primer lugar, están distribuidos de forma casi homogénea por todo el genoma y, en segundo lugar, presentan un número elevado de alelos con frecuencias similares entre sí, de forma que la probabilidad de que un individuo sea heterocigoto es muy elevada. El genoma humano contiene al menos 30.000 microsatélites localizados en la eucromatina.
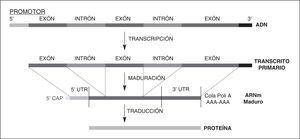
El tipo más frecuente de PG es el de un solo nucleótido, o diferencias de una sola base que aparecen en la secuencia del ADN entre individuos de una población. Se estima que 1 de cada 200-300 bases del ADN humano podría ser un SNP2. Los SNP pueden ocurrir tanto en regiones codificadoras (exones) como en no-codificadoras (intrones y promotores) del genoma (fig. 1). Debido a que sólo el 3%-5% del ADN humano codifica para la producción de proteínas, la mayoría de los SNP se encuentran fuera de las regiones codificadoras. Cuando el SNP ocurre dentro de la región codificadora del gen, o exón, la probabilidad de que se altere la función biológica de la proteína es mayor, ya que el cambio de base puede traducirse en la sustitución de un aminoácido por otro.
Figura 1. Estructura de un gen, transcripción y traducción a proteína.
Mucho más frecuentemente, los SNP ocurren en regiones del gen que no se traducen en proteína. Sin embargo, estos SNP también pueden tener efectos biológicos. Por ejemplo, un SNP en la región promotora de un gen (parte de un gen que contiene la información necesaria para activarlo o desactivarlo) puede alterar la afinidad por la unión con factores de transcripción, o alterar la actividad enhancer (potente intensificador de la expresión de un gen), variando de esta forma los niveles de transcripción del gen y produciéndose, como consecuencia, cambios en los niveles de proteína. Los SNP en las regiones 5'UTR y 3'UTR (regiones 5' y 3' del ARNm, no traducidas) pueden alterar la estabilidad del ARN mensajero (ARNm), afectando su traducción a proteína. Parece menos probable que los SNP presentes en intrones (región no codificadora de un gen) también tengan un significado funcional. Es interesante señalar que los SNP situados en intrones son los más frecuentes, seguidos de los que se producen en regiones promotoras, siendo los menos frecuentes los SNP que afectan a la secuencia de aminoácidos, lo que refleja una gran presión selectiva que limita los cambios en las proteínas. También hay una enorme diversidad en la frecuencia de SNP entre genes, reflejando diferentes presiones selectivas en cada gen, así como diferencias en los niveles de mutación y recombinación a lo largo del genoma. Otro hecho muy interesante es el desequilibrio de ligamiento (linkage disequilibrium) que es el fenómeno por el que dos alelos en diferentes loci aparecen juntos más frecuentemente de lo que cabría esperar por probabilidad.
En relación con las diferencias en las clases de cambio de base en el ADN, las transiciones (púrica → púrica/pirimidínica → pirimidínica) son mucho más frecuentes que las transversiones (púrica → pirimidínica/pirimidínica → púrica). Además, los alelos G (guanina) y C (citosina) tienden a ser los alelos más frecuentes, mientras que A (adenina) y T (timina) son generalmente los alelos menores2. La terminología aquí empleada se resume en la tabla 1.
Métodos para genotipar polimorfismos genéticos
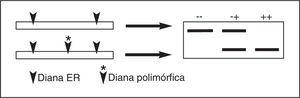
Los SNP pueden ser genotipados mediante la técnica de polimorfismos del tamaño de los fragmentos de restricción (RFLP del inglés, restriction fragment length polymorphism), mediante extensión del cebador, técnica que mide la capacidad del ADN polimerasa para extender un oligonucleótido a través de un sitio polimórfico en la presencia de nucleótidos que sólo permiten la extensión de una de las variantes, o mediante secuenciación del ADN. La primera de estas técnicas es la más utilizada por su facilidad de realización, si bien es una técnica que tiene como inconveniente el estar sujeta a cierta subjetividad. La RFLP se basa en que el cambio de un nucleótido altera la diana para una determinada enzima de restricción (ER) con lo que podemos detectar el polimorfismo en función de la variación que éste genera en el patrón de restricción. Para ello se amplifica el ADN con sondas específicas mediante reacción en cadena de polimerasa (PCR), se somete a restricción enzimática y posteriormente se realiza una electroforesis (fig. 2).
Figura 2. Polimorfismos del tamaño de fragmentos de restricción (RFLP). La pérdida o ganancia de una diana de restricción por efecto de la sustitución de un nucleótido puede ser detectado por las variaciones en los tamaños de los fragmentos de restricción. En la figura se presenta el resultado esperado de este tipo de polimorfismos en individuos homocigotos para la presencia de la diana (+ +), los homocigotos para la ausencia de diana (- -) y los heterocigotos (+ -). ER: enzima de restricción.
Los polimorfismos de repetición pueden ser genotipados mediante PCR, utilizando como cebadores oligonucleótidos que flanquean a la región que contiene la repetición. De cada individuo se amplifican fragmentos de ADN que difieren entre sí por el número de repeticiones. El resultado de la amplificación se fracciona en un gel de acrilamida para determinar el genotipo de cada individuo.
POLIMORFISMOS GENÉTICOS DE MOLÉCULAS Y RECEPTORES ENCARGADOS DE RECONOCER LA ENDOTOXINA
Proteína ligadora de lipopolisacárido
Polimorfismos de la proteína ligadora de lipopolisacárido
El gen que codifica la proteína ligadora de lipopolisacárido (LBP) se localiza en el cromosoma 20. Se han descrito en el exón de este gen dos SNP: en posición 291 en la que se sustituye un residuo de T por G y en posición 1306 en la que se cambia un residuo de C por T. Ambos polimorfismos conllevan un cambio en la secuencia de aminoácidos.
Polimorfismos de la proteína ligadora de lipopolisacárido, sepsis y mortalidad
Al estudiar una cohorte de 204 pacientes con sepsis, el poseer un residuo G en posición 292 incrementaba el riesgo de sepsis al comparar con el grupo control, si bien no afectaba a la mortalidad de los pacientes con sepsis3.
Receptor CD14
Polimorfismos del receptor CD14
El gen que codifica el receptor CD14 se localiza en el cromosoma 5. Se ha identificado un polimorfismo situado en posición -260, la región promotora del gen que codifica este receptor consistente en la sustitución de C por T.
Polimorfismos del receptor CD14, sepsis y mortalidad
Un estudio realizado en 90 pacientes con shock séptico ha demostrado que el genotipo TT de este polimorfismo es más frecuente que en el grupo control y que la mortalidad de estos sujetos es significativamente superior que en pacientes con otros genotipos. En el análisis multivariante controlando con otros factores de confusión, la edad (1,04; intervalo de confianza 95%, 1,01-1,08) y el genotipo TT (5,30; intervalo de confianza 95%, 1,20-22,50) se asociaron de forma independiente con un incremento del riesgo de muerte en Unidades de Cuidados Intensivos (UCI)4.
Polimorfismos del CD14 y desarrollo de sepsis
Hensen et al no han hallado influencia de este genotipo en el desarrollo de sepsis al evaluar 58 pacientes traumatizados con injury severity score (ISS) superior a 165.
POLIMORFISMOS GENÉTICOS DE CITOCINAS PROINFLAMATORIAS
Factor de necrosis tumoral
El factor de necrosis tumoral (tumor necrosis factor, TNF) es la primera citocina que se libera tras la agresión de un agente infeccioso o de otro tipo. Es un activador de la inmunidad innata y favorece la acción de las células fagocíticas.
Polimorfismos del factor de necrosis tumoral
Diversos polimorfismos han sido identificados en el gen que codifica el TNF y que se encuentra en el brazo corto del cromosoma 6, lo cual explicaría que la producción de TNF varíe de un paciente a otro. De todos ellos, dos son de especial interés y han sido ampliamente estudiados en el paciente crítico:
1. -308 (G/A) polimorfismo: en la región promotora del gen que codifica el TNF-α, en la posición -308, es decir, 308 nucleótidos antes de que empiece la región transcriptora, existe una mutación descrita. Normalmente, este lugar está ocupado por un residuo de G pero en un 20% está ocupado por A. Dado que existen dos copias en el cromosoma, esta mutación puede ser G/G (80% de los casos), G/A (15%) o A/A (5%). Las copias que contienen G se denominan como alelo TNF1 y las que poseen A como TNF2. La presencia del alelo TNF2 se asocia con una mayor producción in vitro de TNF-α. Existen otros polimorfismos descritos en esta región promotora pero no se traducen en general en modificaciones en la producción del TNF-α y han sido escasamente estudiados en el paciente crítico.
2. El TNF-β, -Ncol polimorfismo: cerca de la región donde se codifica el TNF-α, se encuentra el gen para el TNF-ß. El primer intrón (región de ADN que separa genes pero no codifica) de este gen posee un residuo A en posición 1069 (TNFB2) pero puede contener G (TNFB1). Las frecuencias de genotipos son: 10% para G/G, 50% para G/A y 40% para A/A. La variante G (TNFB1) se caracteriza porque es cortada específicamente por una enzima de restricción NcoI. Este polimorfismo conlleva un cambio de aminoácido en la posición 26 (asparagina para TNFB1 y treonina para TNFB2). Los sujetos homocigóticos TNFB1 presentan una mayor producción de TNF-ß mientras que los homocigóticos para TNFB2 presentan mayor producción de TNF-α.
Polimorfismos del factor de necrosis tumoral, sepsis y mortalidad
Han sido publicados resultados contradictorios en cuanto a la repercusión sobre el pronóstico de estos dos polimorfismos del TNF en los pacientes con sepsis. Así, mientras en una serie de Stuber et al que estudió un grupo de pacientes con sepsis grave el ser TNF2 no se asoció a mayor mortalidad, en un estudio que incluyó 89 pacientes con shock séptico y 87 controles, los pacientes con alelo TNF2 (homocigóticos o heterocigóticos) presentaron mayor incidencia de shock séptico y mortalidad hospitalaria que los pacientes con TNFB1, si bien no hubo diferencias en los niveles circulantes de TNF-α. Tras un análisis multivariante para controlar con otras variables de confusión, los pacientes con el alelo TNF2 tenían un mayor riesgo de muerte (3,7; intervalo de confianza 95%, 1,37-10,24)6.
En cuanto al -Ncol polimorfismo, Stüber et al7 estudiaron el genotipo para TNFB en 40 pacientes con sepsis grave. La distribución en esta serie fue TNFB1 homocigótico 10%, TNFB1/TNFB2 heterocigótico 48% y TNFB2 homocigótico 42%. Aquellos pacientes que fallecieron tenían una más alta prevalencia de TNFB2 (p < 0,005). Los pacientes homocigóticos para TNFB2 tuvieron una más elevada mortalidad que los heterocigóticos (TNFB1/TNFB2); (p = 0,0022), así como niveles circulantes de TNF más elevados. Resultados similares fueron posterioremente confirmados, en cuanto a mayor mortalidad para los homocigóticos de TNFB2, por este grupo al analizar el polimorfismo de los heat shock protein (HSP), proteínas celulares implicadas en los procesos de reparación de diversas proteínas esenciales para la supervivencia celular, y el polimorfismo de otras dos citocinas implicadas en la sepsis como la interleucina 1 (IL-1) y el antagonista del receptor de la IL-1 (IL-1 ra), sin que los polimorfismos de las HSP influyan en el pronóstico8.
Recientemente, un estudio que incluyó neumonías de la comunidad, demostró que los sujetos TNFB2 (AA) tienen mayor probabilidad de desarrollar shock séptico sin que exista influencia en el caso del polimorfismo -308 (G/A). Hay que destacar que el genotipo hiposecretor (TNFB1; GG) se asoció a una mayor frecuencia de fallo respiratorio. Ninguno de los dos polimorfismos del TNF se asoció a una mayor mortalidad9.
Polimorfismos del factor de necrosis tumoral y desarrollo de sepsis
El grupo de Stüber ha evaluado la influencia del -Ncol polimorfismo en el desarrollo de sepsis tras diversas situaciones de agresión. Así, en 110 pacientes con traumatismo grave, la sepsis grave fue significativamente más frecuente en sujetos homocigóticos para el alelo TNFB210.
Interleucina
La IL-1 (IL-1α e IL-1β) es una importante citocina proinflamatoria mientras que IL-1 ra se une al mismo receptor pero no induce activación celular. Los tres genes correspondientes, localizados en el brazo largo del cromosoma 2, se denominan respectivamente IL1A, IL1B e IL1RN.
Polimorfismos de la familia de la interleucina 1
La IL-1α posee 7 alelos según el número de repeticiones en tándem de 46 pares de bases en el intrón 6, la IL-1β posee dos alelos en el exón 5 y el gen del IL-1 ra posee un número variable de repeticiones en tándem de 86 pb en el intrón 2 con 5 alelos. El gen IL1B contiene además, dos SNP en posición -511 y +3593 que influencian la producción in vitro de IL-1.
Polimorfismos de la interleucina 1, sepsis y mortalidad
Un estudio realizado en 93 pacientes con sepsis grave no halló relación entre los polimorfismos de la IL-1ß y del IL-1 ra y la mortalidad confirmando, por otro lado, la asociación entre la mortalidad y el alelo TNFB2 del TNFß Ncol polimorfismo. En este estudio se observó que los pacientes con sepsis tenían una frecuencia significativamente superior del alelo RN2 respecto al grupo control.
Por contra, un estudio realizado en 78 pacientes con sepsis grave observó influencia del polimorfismo genético en el intrón 2 del gen IL-1 ra en el pronóstico de la sepsis. El riesgo de fallecer en el análisis multivariante fue significativamente superior para los pacientes homocigóticos para el alelo RN2 (6,47; intervalo de confianza 95%, 1,01-41,47)11.
Interleucina 6
La IL-6 es una citocina mayoritariamente proinflamatoria que es sintetizada principalmente por el monocito/macrófago. Induce la síntesis de reactantes de fase aguda a nivel hepático.
Polimorfismos de la interleucina 6
Aunque han sido diversos polimorfismos en la región promotora del gen de la IL-6 que se encuentra en el cromosoma 7, sólo el localizado en la posición -174 ha sido evaluado en diversas situaciones clínicas. En esta posición existe un SNP que consiste en el cambio de una G por C. El alelo G se asocia con niveles más elevados de IL-6 en adultos sanos.
Polimorfismos de la interleucina 6, sepsis y mortalidad
En un estudio realizado en pacientes quirúrgicos, la mortalidad en la sepsis fue significativamente inferior en los sujetos homocigóticos GG con un riesgo relativo comparado con otros genotipos de 0,11 (intervalo de confianza 95%, 0,02-0,57). En este estudio, los homocigóticos GG no presentaban niveles séricos más elevados de IL-6 pero sí los pacientes que fallecieron tenían niveles séricos de IL-6 significativamente superiores que los que sobrevivieron. Los autores especulan que pueden existir otros polimorfismos genéticamente relacionados que expliquen estos hallazgos12.
POLIMORFISMOS GENÉTICOS DE CITOCINAS ANTIINFLAMATORIAS
Interleucina 10
La IL-10 junto con la IL-4 son las citocinas antiinflamatorias más potentes. La IL-10 es sintetizada por los linfocitos Th2 e inhibe la producción de citocinas proinflamatorias por parte del monocito/macrófago.
Polimorfismos de la interleucina 10
El gen que codifica la IL-10 se encuentra en el cromosoma 1, habiéndose descrito diversos polimorfismos en la región promotora: -1082 (G/A), -819 (C/T) y -592 (C/A), si bien se ha demostrado que los polimorfismos en los pb -592 y -819 están en completo desequilibrio de ligamiento. En cuanto a la traducción de estos cambios se han obtenido resultados contradictorios. Así, se ha comprobado que en la mutación en posición -1082, el cambio de G por A conlleva modificaciones en la síntesis de esta citocina ya que en los pacientes homocigóticos GG la producción es significativamente superior que en los sujetos homocigóticos que poseen un residuo de A en dicha posición13. Por el contrario, recientemente, se ha observado que este cambio de pares de base en -1082 no se traduce en diferencias en la producción in vitro de IL-10 mientras que los sujetos homocigóticos CC en posición -592 tienen la más elevada liberación de esta citocina14.
Polimorfismos de la interleucina 10, sepsis y mortalidad
En cuanto a la traducción clínica de estas mutaciones, estudios recientes han mostrado conclusiones contradictorias estudiando grupos heterogéneos de pacientes críticos. Así, Reid et al15 sólo mostraron que el genotipo hiperproductor de IL-10 era significativamente menos frecuente en los pacientes que ingresaban en UCI con síndrome de disfunción multiorgánica (SDMO) respecto a un grupo control sin que ningún genotipo de la IL-10 se asociara a mayor mortalidad.
Como hemos visto a lo largo de esta revisión, se han evaluado múltiples polimorfismos en pacientes críticos obteniéndose resultados no siempre coincidentes e incluso en ocasiones contradictorios. Los factores que pueden explicar este hecho son múltiples:
1. Momento de inclusión en los estudios: el momento de identificación de los pacientes es extremadamente importante para obtener conclusiones válidas sobre un determinado factor pronóstico.
2. Tamaños muestrales inadecuados: la patogenia de la sepsis es muy compleja, estando implicados múltiples mediadores cuya función no siempre conocemos bien y que puede variar evolutivamente.
3. Elección de la variable clínica a analizar: esta variable debe ser objetiva para evitar sesgos de interpretación. Por ello se escoge habitualmente la mortalidad hospitalaria si bien esto plantea dudas de si otras variables serían más adecuadas para poner de relieve la traducción clínica en la modificación de un gen determinado.
4. Inclusión en estudios de pacientes de diversas etnias.
5. Errores a la hora de determinar el polimorfismo genético como puede ocurrir al determinar los pacientes heterocigotos del SNP en posición -308 del TNF mediante enzima de restricción y electroforesis. Este caso puede deberse a una digestión incompleta por parte de la enzima de restricción, por lo que podría ser conveniente prolongar el tiempo de incubación con la enzima.