Un diagnóstico y tratamiento precoz tiene un impacto importante en la morbimortalidad de las infecciones producidas por bacterias multirresistentes. Los bacilos gramnegativos multirresistentes constituyen la principal amenaza actual en los hospitales y muy especialmente en las unidades de cuidados intensivos. El papel del laboratorio de microbiología es esencial en dar una respuesta rápida y eficaz. En esta revisión se actualiza los procedimientos del laboratorio de microbiología para la detección rápida de los bacilos gramnegativos multirresistentes y de sus determinantes de resistencia. También se estudia el papel del laboratorio en la vigilancia y control de brotes por estas bacterias, incluyendo las técnicas de tipificación. Se destaca la importancia de proporcionar mapas de resistencia normalizados que permitan conocer la situación epidemiológica de las diferentes unidades. Finalmente se revisa la importancia de sistemas de comunicación eficaces para la transmisión de resultados y la toma de decisiones en el manejo de pacientes infectados por bacilos gramnegativos multirresistentes.
Early diagnosis and treatment has an important impact on the morbidity and mortality of infections caused by multidrug-resistant bacteria. Multidrug-resistant gram-negative bacilli constitute the main current threat in hospitals and especially in intensive care units. The role of the microbiology laboratory is essential in providing a rapid and effective response. This review updates the microbiology laboratory procedures for the rapid detection of multidrug-resistant gram-negative bacilli and its resistance determinants. The role of the laboratory in the surveillance and control of outbreaks caused by these bacteria, including typing techniques, is also studied. The importance of providing standardized resistance maps that allow knowing the epidemiological situation of the different units is emphasized. Finally, the importance of effective communication systems for the transmission of results and decision making in the management of patients infected by multidrug-resistant gram-negative bacilli is reviewed.
La resistencia antimicrobiana es un problema de salud pública mundial. El número de infecciones por bacterias multirresistentes aumenta cada año en numerosos centros y no se circunscriben al ámbito hospitalario, sino también a la comunidad1. Existen numerosas evidencias que demuestran que el tratamiento adecuado y precoz de las infecciones graves por bacterias multirresistentes disminuye la morbilidad, la mortalidad y la estancia hospitalaria2. Este hecho es de especial relevancia en las unidades de cuidados intensivos (UCI), donde los factores de riesgo para adquirir este tipo de infecciones son especialmente relevantes. Las infecciones más relevantes en la actualidad son las producidas por bacterias gramnegativas, destacando las enterobacterales, particularmente Klebsiella pneumoniae K. pneumoniae), productoras de betalactamasas de espectro extendido (E-BLEE) y/o carbapenemasas (EPC), Pseudomonas aeruginosa (P. aeruginosa) y Acinetobacter baumannii(A. baumannii) resistentes a los carbapenémicos. Además, estas bacterias multirresistentes pueden pertenecer a clones de alto riesgo, que suman a su multirresistencia, una gran capacidad de diseminación y de colonización de pacientes o superficies ambientales3. Estas bacterias, resistentes a casi todos los betalactámicos, lo son además a otras familias de antimicrobianos, tales como quinolonas o aminoglucósidos. En algunos casos, existen pocas alternativas terapéuticas para el tratamiento de las infecciones producidas por estos microorganismos. El uso empírico de tratamientos antimicrobianos de amplio espectro puede favorecer el desarrollo de las resistencias en las UCI. Para evitar esto es necesario disponer con rapidez de datos sobre la identificación, mecanismos de resistencia y sensibilidad antimicrobiana de los microorganimos causantes de la infección. En este sentido, el papel del laboratorio de microbiología es esencial. Debe tener la capacidad de suministrar la información necesaria sobre la etiología y sensibilidad/resistencia antimicrobiana en el menor tiempo posible de tal manera que permita el uso de tratamientos dirigidos adecuados que mejoren el pronóstico de los pacientes y que tenga el menor impacto posible en el fenómeno de la resistencia antimicrobiana4,5. Los métodos y técnicas utilizadas se deben adaptar a la realidad epidemiológica de cada centro, pero el suministro de esta información con rapidez es especialmente relevante en unidades con infecciones graves o en pacientes con importantes enfermedades de base, como ocurre en las UCI. Para desarrollar estas actividades, es esencial disponer de accesibilidad a los servicios de microbiología 24 h al día, 7 días de la semana. En este artículo vamos a revisar el papel de los laboratorios de microbiología en el diagnóstico y tratamiento de las infecciones por bacilos gramnegativos multirresistentes (BGN-MR), así como para la vigilancia y control de las infecciones producidas por estos microorganismos en las UCI.
Diagnóstico de infecciones por BGN-MR. Recomendaciones de toma de muestras y significado de los hallazgos microbiológicosLas infecciones por BGN-MR en pacientes graves ingresados en la UCI están principalmente asociadas a ventilación mecánica, bacteriemia asociada a catéter, infección quirúrgica e infección urinaria, aunque cualquier órgano o tejido puede ser infectado6. Estos microorganismos inicialmente colonizan piel y mucosas de los pacientes, adquiriéndolos bien a partir de fómites, manos del personal o de superficies colonizadas de la UCI, o bien tras la selección con antibióticos de la flora intestinal del propio paciente. La colonización del paciente aumenta el riesgo de desarrollar una infección posterior por estos microrganismo7,8. El caso de EPC puede llegar a ser del 17% de los colonizados9. El grado elevado de colonización de los pacientes de UCI dificulta una correcta interpretación de los hallazgos microbiológicos. El cultivo de BGN-MR en muestras estériles como sangre, líquido pleural o aspirados casi siempre implican infección. En cambio, el aislamiento en muestras respiratorias, orina en pacientes sondados o muestras de heridas abiertas requieren de una correlación clínica para diferenciar colonización de infección. En este sentido, los cultivos cuantitativos de muestras respiratorias10 y de orina11 mejoran la especificidad, utilizando puntos de corte para estas muestras (tabla 1). La tinción de Gram de estas muestras puede aportar información que sugiera contaminación y para el diagnóstico precoz de la neumonía.
Valoración microbiológica del cultivo de muestras
| Tipo de muestra | Punto de corte | Parámetros adicionales que valorar |
|---|---|---|
| Muestras respiratorias | ||
| Lavado broncoalveolar | ≥ 104 UFC/mL | ≥ 5% células con bacterias intracelulares |
| Cepillo protegido | ≥ 103 UFC/mL | |
| Aspirado endotraqueal | ≥ 106 UFC/mL | <1% de células epiteliales |
| Muestras de orina | ||
| Orina por sondaje vesical | ≥100 UFC/mL | |
| Sonda permanente | ≥1000 UFC/mL | Leucocitos en orina |
Los métodos más usados basados en medios de cultivo tradicionales son (tabla 2):
Características de técnicas para detección rápida de resistencia en BGN-MR
| Característica relevante | Medios selectivos/cromogénicos | MALDI-TOF | Inmunocromatografía | Colorimétricos | Accelerate Pheno | rAST-EUCAST | Alfred 60 AST |
|---|---|---|---|---|---|---|---|
| Facilidad de uso | +++ | ++ | +++ | +++ | ++ | +++ | ++ |
| Interpretación de resultados | Crecimiento vs. no crecimiento | Análisis de picos en el espectro | Líneas visibles en una membrana para cada tipo de carbapenemasa | Cambio de color | Interpretación por el software S/R | Zonas de inhibición alrededor de discos tras incubación | Análisis de curvas de crecimiento |
| Tiempo de resultado | 24 h | 4 h | 15 min | 30 min-2 h | 8 h | 4-8 h | 4-6 h |
| Coste | + | ++ | ++ | ++ | ++++ | + | +++ |
1. Medios de cultivo selectivos. Pueden prepararse en el laboratorio o pueden utilizarse medios comerciales. Algunos medios comerciales presentan la ventaja de incluir sustratos cromogénicos incoloros que, debido a la actividad enzimática específica de cada microorganismo, proporcionan a la colonia un color intenso y específico que permite la identificación presuntiva. Estos medios se deben utilizar como método de cribado y siempre debe confirmarse especie y mecanismo de resistencia con otros métodos12,13.
2. Método de detección de carbapenemasas basado en la difusión con discos de inhibidores de carbapenemasas, BLEE y AmpC14.
MALDI-TOF es un sistema de espectrometría de masas que permite la identificación rápida de bacterias mediante el análisis de proteínas. También puede ser útil para detectar actividad carbapenemasa mediante la detección de la hidrólisis de carbapenémicos15,16. Esta técnica puede ser coste-efectiva si se dispone del equipo, aunque precisa un módulo especial de análisis, preparación de reactivos y personal formado para su realización e interpretación. Los reactivos suelen prepararse en el laboratorio, aunque hay kits comerciales como MBT STAR-Carba (Bruker Daltonics) que detectan carbapenemasas17,18. Este test no pone de manifiesto otros posibles mecanismos de resistencia a carbapenémicos ni tampoco identifica el tipo de carbapenemasa. Una nueva aplicación de este sistema, aún en fase experimental, es la detección de proteínas asociadas a plásmidos portadores de carbapenemasas19.
La inmunocromatografía es una técnica sencilla de realizar y de interpretar, basada en la utilización de anticuerpos específicos del tipo de carbapenemasa.
Uno de los métodos más utilizados es el NG-Test Carba 5 que detecta las principales carbapenemasas (KPC, OXA-48, NDM, IMP, VIM), aunque se han descrito falsos positivos (100% sensibilidad; >95% especificidad). Producen resultados en 15 min, incluso con aislados procedentes de hemocultivo positivo20,21. Recientemente se ha adaptado esta tecnología para la detección de BLEE de tipo CTX-M22.
Los métodos colorimétricos rápidos detectan la presencia de enzimas (carbapenemasas, BLEE) mediante hidrólisis de sustratos que producen una modificación del pH y un cambio de color. Actualmente se dispone de varios test comerciales como RAPIDEC CARBA NP (Biomerieux), β-CARBA (Bio-Rad), β-LACTA (Bio-Rad), MAST PAcE (Mast Diagnostics). Algunos de estos test presentan baja sensibilidad para detectar enzimas de tipo OXA, y en la detección de carbapenemasas infrecuentes23,24.
Finalmente, existen métodos de antibiograma rápidos que no se centran en la detección de un solo fenotipo de resistencia, sino que permiten obtener información de más antimicrobianos.
Accelerate Pheno System (Accelerate Diagnostics)Es un sistema automatizado de identificación y antibiograma de muestras positivas de hemocultivo en aproximadamente 8h. A través de un análisis morfocinético el sistema analiza características fenotípicas como tamaño, forma y tasa de división de las bacterias que crecen en microcolonias en presencia del antibiótico. La concordancia en la identificación oscila entre 87,9-100% y en la categoría clínica es mayor del 91%25. Los principales inconvenientes son su coste, solo determina la sensibilidad de las especies incluidas en su panel de identificación, y su menor rendimiento en infecciones polimicrobianas. Se han realizado estudios utilizando líquidos estériles cultivados en frascos de hemocultivo pero el rendimiento es menor debido a la mayor probabilidad de incluir infecciones polimicrobianas o producidas por microorganismos no incluidos en el panel25,26.
Antibiograma de lectura rápida siguiendo criterios de EUCAST (rAST-EUCAST)EUCAST ha validado un método sencillo y barato para realizar una lectura rápida (4, 6 u 8 h de incubación) del antibiograma utilizando el método de difusión con discos tras inocular las placas directamente de un frasco de hemocultivo positivo27,28. Es un método estandarizado y actualmente solo se ha validado para 8 especies (Escherichia coli, K. pneumoniae, P. aeruginosa, Staphylococcus aureus, Streptococcus pneumoniae, Enterococcus faecalis, Enterococcus faecium y A. baumannii), lo cual requiere una identificación rápida del microorganismo para poder seleccionar el antibiograma. Uno de sus inconvenientes es que si no se obtienen resultados fiables tras 8 h es necesario realizar un antibiograma convencional. Se han realizado estudios comparando rAST-EUCAST con otros métodos y se ha comprobado que existe buena concordancia29.
Alfred 60 AST (Alifax)Este sistema proporciona un antibiograma directamente a partir de frascos de hemocultivos positivos en 4-6h. El sistema utiliza la medida de la dispersión de la luz para detectar crecimiento bacteriano en medio líquido con antibióticos calculando un porcentaje de inhibición de crecimiento frente al crecimiento en medio sin antibiótico. Este método no realiza identificación bacteriana, por lo cual se requiere una identificación rápida previa. Estudios comparativos con otras técnicas han mostrado una concordancia entre 90-95%30,31.
Actualmente se encuentran en fase de desarrollo y comercialización nuevas técnicas basadas en microfluidos, biosensores, óptica, electrónica y amplificación de ADN, que supondrán una revolución en la detección de la resistencia a antimicrobianos32.
Técnicas molecularesLa detección de BGN-MR en el laboratorio de microbiología requiere una correcta identificación de la especie bacteriana y de los determinantes de resistencia. Los métodos moleculares más utilizados para identificar BGN se basan en la detección de proteínas (métodos de proteómica), como MALDI-TOFF, que identifica correctamente la mayoría de las bacterias multirresistentes patógenas, aunque presenta problemas de especificidad para detectar los mecanismos de resistencia más relevantes, y son relativamente laboriosos33. Existe una gran diversidad de técnicas moleculares para identificar los determinantes de resistencia, siendo las más utilizadas las basadas en la detección de ácidos nucleicos (AN) (métodos genéticos), que tienen una elevada sensibilidad y especificidad, y se pueden utilizar a partir de colonia o de muestra directa34. Los métodos moleculares genéticos más utilizados se basan en reacciones de amplificación génica y detección de amplicones. El método de amplificación más utilizado en la detección de BGN-MR es la reacción en cadena de la polimerasa (PCR), aunque en los últimos años se están utilizando cada vez con más frecuencia otras alternativas de menor coste y más rápidas, como las técnica de amplificación isotérmica (ej. LAMP)35. Existen 3 grandes grupos de técnicas de amplificación de AN en tiempo real: técnicas basadas en la amplificación de la diana (ej. PCR, NASBA, etc.), técnicas de amplificación de la señal (ej, bDNA, captura de híbridos) y técnicas de amplificación de la sonda (LCR)36. La técnica de amplificación más conocida es la PCR, tanto en su formato convencional (PCR simple, PCR anidada, PCR múltiple, RT-PCR) como en tiempo real35. La detección de amplicones puede realizarse por electroforesis en gel de agarosa y tinción con bromuro de etidio (PCR convencional), mediante hibridación con sondas fluorescentes específicas marcadas (ej., sondas Taqman, sondas FRET, sondas molecular beacons, sondas escorpions, etc.) o por técnicas de secuenciación de AN33,35.
La secuenciación de AN permite identificar genes que codifican determinantes de resistencia antimicrobiana, genes reguladores de dichos determinantes de resistencia, así como los elementos genéticos móviles, como plásmidos y transposones que diseminan o movilizan estos determinantes de resistencia.
Los secuenciadores de AN que se utilizan habitualmente se basan en el método enzimático de terminación de cadena de Sanger (secuenciación poblacional), aunque en los últimos años se han desarrollado nuevas tecnologías o plataformas para secuenciación masiva (secuenciación clonal) o de alto rendimiento (next-generation sequencing), como la de Illumina, PacBio o Nanopore, que permiten secuenciar genomas bacterianos completos37,38. La secuenciación masiva permite además obtener información útil para la tipificación (ver apartado de tipificación molecular) o para el estudio de genes de virulencia de BGN-MR37.
Las principales ventajas de las técnicas moleculares en comparación con las técnicas convencionales (cultivo, etc.) son su rapidez y su mayor sensibilidad analítica, por lo que algunas de ellas, como la LAMP, se utilizan como técnicas rápidas para la detección de BGN-MR (point of care), que son especialmente útiles cuando se aplican a paneles sindrómicos, pues permiten detectar múltiples patógenos multirresistentes y mecanismos de resistencia antimicrobiana39,40. Los paneles de sepsis pueden identificar un gran número de bacterias multirresistentes y mecanismos de resistencia y carbapenemasas a partir de frascos de hemocultivos positivos o de muestras de sangre con EDTA (tabla 3). Muchas de estas técnicas están automatizadas y comercializadas y permiten trabajar con un elevado número de muestras, lo que facilita el trabajo técnico y redunda en la calidad de los resultados.
Sistemas de identificación de patógenos productores de sepsis, y de determinantes de resistencia aprobados por la FDA
| Característica | Sistema o plataforma | |||||
|---|---|---|---|---|---|---|
| BioFire FilmArray(Biomerieux) | Verigene (Luminex) | ePlex BCID (GenMark) | Unyvero (Curetis) | Sepsis Flow Chip Assay (Master) | Magicplex Sepsis (Seegene) | |
| Tecnología | PCR multiplex | PCR + Microarray | PCR + Microarray | PCR + Microarray | PCR + Microarray | PCR convencional y RT-PCR |
| Muestra clínica | ||||||
| Hemocultivo positivo | X | X | X | X | X | |
| Sangre directa | X | |||||
| Tinción de Gram | X | X | ||||
| Tiempo respuesta (h) | 1 | 2.5 | 1.5 | 4 | 3 | 6 |
| Tiempo manipulación manual (min) | <2 | 10 | <2 | <2 | NC | NC |
| Patógenos detectados | 24 | 22 | 57 | 35 | 18 | 91 |
| Gramnegativos | 11 | Panel GN (9) | Panel GN(21) | Panel GN (15) | 10 | 12 |
| Grampositivos | 8 | Panel GP: 13 | Panel GP: 20 | Panel GP: 11 | 7 | 73 |
| Hongos | Candida spp. (5) | Panel hongos(16) | Panel hongos(8) | C. albicans (1) | Hongos(6) | |
| M. tuberculosis | X | |||||
| N.° de determinantes de resistencia | 3 | 8 | 8 | 16 | 20 | 3 |
| mecA | X | X | X | X | X | X |
| VanA/VanB | X | X | X | X | X | X |
| CTX-M | X | X | X | X | X | |
| OXA-48 | X | X | X | |||
| KPC | X | X | X | X | X | |
| VIM | X | X | X | X | ||
| NDM | X | X | X | X | ||
| IMP | X | X | X | X | ||
Las limitaciones más relevantes de las técnicas moleculares genómicas son su elevado coste y la necesidad, en algunos casos, de personal con experiencia en este tipo de técnicas y de herramientas bioinformáticas para analizar la información obtenida.
Interpretación del antibiograma. Algoritmos de caracterización de resistenciaEn la lectura de un antibiograma se categorizan (S/I/R) los resultados cuantitativos obtenidos de la evaluación de la actividad de un antimicrobiano frente a un microorganismo realizada mediante métodos estandarizados. En los años 90, se fue introduciendo el concepto de lectura interpretada del antibiograma41. Este concepto implica un análisis fenotípico completo de los resultados obtenidos frente a todos los antibióticos para poder inferir posibles mecanismos de resistencia basados en el conocimiento de cómo se expresan fenotípicamente42,43. La lectura interpretada es una labor compleja para el microbiólogo ya que, aunque existen sistemas automatizados con reglas experto, la resistencia microbiana está en continua evolución y es preciso una actualización de conocimientos continua. Como ejemplo de lectura interpretada en BGN-MR, es fundamental analizar de manera conjunta todos los antibióticos betalactámicos ya que es la base de la detección de BLEEs y carbapenemasas, que destacan por su especial importancia epidemiológica.
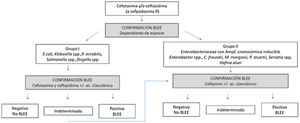
BLEE. En enterobacterales se han descrito infinidad de enzimas de este tipo (CTX-M, SHV, TEM). Las propiedades de cada enzima, sus niveles de expresión y la presencia de otros posibles mecanismos añadidos (alteraciones de la permeabilidad de membrana, bombas de expulsión activa del antibiótico localizadas en la membrana o la presencia de otras betalactamasas) hacen posible una amplia diversidad de fenotipos44. Debe sospecharse la presencia de BLEE cuando la CMI de cefotaxima/ceftriaxona y/o ceftazidima es>1mg/l (cefpodoxima>1mg/l es un indicador más sensible pero menos específico). Siempre deben ensayarse cefotaxima/ceftriaxona y ceftazidima en paralelo ya que puede haber diferencias en los valores de CMI según el tipo de BLEE. Un valor de CMI para cefepime>4mg/l, especialmente en E. coli y K. pneumoniae, apoya la presencia de una BLEE como mecanismo subyacente. Cuando tras la lectura se sospecha que puede existir una BLEE debe realizarse una prueba de confirmación, basado en la inhibición in vitro de la actividad de la betalactamasa por ácido clavulánico14. En la figura 1, se muestra un ejemplo de algoritmo de confirmación de presencia de BLEE según EUCAST.
Carbapenemasas. Al igual que ocurre con las BLEE, es posible observar múltiples fenotipos debido a la gran variabilidad de carbapenemasas descritas45. Es importante recordar también que la resistencia a carbapenémicos puede estar mediada por la combinación de mecanismos distintos de las carbapenemasas (ej. pérdida de porinas en cepas hiperproductoras de AmpC). En las enterobacterales productoras de carbapenemasa los valores de CMI de los carbapenémicos pueden ser inferiores a los puntos de corte clínicos, por lo que se han establecido puntos de corte más bajos (CMI>0,125mg/l para meropenem o ertapenem, teniendo este último mayor sensibilidad pero menor especificidad) para la detección de carbapenemasa. Sin embargo, en P. aeruginosa hay que tener en cuenta que en la mayor parte de los casos la resistencia a carbapenémicos se debe a la combinación de mecanismos intrínsecos o cromosómicos (alteración de la porina OprD junto con hiperexpresión de bombas de eflujo), aunque la resistencia al resto de betalactámicos, especialmente a la combinación ceftolozano-tazobactam obliga a descartar la presencia de una carbapenemasa46. En A. baumannnii la resistencia a carbapenémicos es mayoritariamente debida a la presencia de una carbapenemasa adquirida de tipo OXA, aunque otras carbapenemasas pueden estar presentes, lo cual debe confirmarse por métodos genotípicos47.
Técnicas de tipificación molecularLas técnicas de tipificación molecular son útiles en la vigilancia y control de brotes causados por BGN-MR porque permiten conocer la clonalidad entre aislados, identificar reservorios y determinar vías de transmisión. Las principales técnicas de tipificación molecular se basan en la electroforesis en campo pulsado (PFGE), la amplificación de AN mediante PCR y la secuenciación de AN48,49. La PFGE es la técnica de referencia de tipificación molecular debido a que tiene un elevado poder de discriminación50. Entre las limitaciones más importantes de la PFGE están el elevado coste del equipo, su laboriosidad, el tiempo necesario para analizar los pulsotipos, la dificultad para diferenciar algunos pulsotipos con bandas de ADN muy próximas, y que existen cepas bacterianas cuyos genomas no se digieren con las enzimas de restricción utilizadas. Aunque hay numerosas técnicas de tipificación basadas en la PCR (tabla 4), la más utilizada es la PCR de secuencias de ADN repetidas (REP-PCR)48,49. Estas técnicas son más económicas, menos laboriosas (algunas son semiautomatizadas y están comercializadas, como Diversilab de bioMérieux), y suelen generar patrones de bandas relativamente más fáciles de interpretar que la PFGE. El poder de discriminación de las técnicas de PCR puede ser inferior al de la PFGE, particularmente en algunos enterobacterales como E. coli y K. pmeumoniae. Tanto la PFGE como las técnicas de PCR tienen que optimizarse o estandarizarse en el laboratorio para garantizar una buena reproducibilidad. Para el estudio de brotes pequeños y de corta evolución, en los que interesa obtener una información con rapidez, podría plantearse la utilización de una técnica de PCR. En brotes más complejos y de mayor duración, en los que interesa conocer además la evolución o dinámica de los clones con el tiempo y su distribución geográfica, es más rentable utilizar la PFGE.
Características más relevantes de algunos métodos moleculares de tipificación de BGN MDR
| Característica | PFGE | Rep-PCR | MLST | SNP |
|---|---|---|---|---|
| Reproducibilidad | Excelente | Buena-excelente | Excelente | Excelente |
| Poder discriminatorio | Excelente | Moderado-excelente | Moderado | Excelente |
| Facilidad interpretación datos | Moderado | Moderado-bueno | Bueno | Bueno |
| Facilidad de uso | Moderado | Bueno | Moderado | Moderado |
| Alto rendimiento | No | Sí | Sí | Si |
| Coste | Moderado | Bajo-moderado | Moderado | Moderado-Alto |
| Tiempo requerido (días) | 3 | 1 | 3-4 | 5-10 |
Los métodos de tipificación basados en la secuenciación de AN pueden basarse en la secuenciación parcial de algunos genes mediante secuenciación tipo Sanger o en la secuenciación de genomas completos mediante técnicas de secuenciación masiva de alto rendimiento51. El ejemplo más representativo de secuenciación Sanger aplicada al tipado molecular es el MLST, basado en la obtención de secuenciotipos (ST) mediante la amplificación y secuenciación parcial de 6-7 genes conservados52. Esta metodología tiene un poder de discriminación inferior al de la PFGE, por lo que suele aplicarse en estudios epidemiológicos globales o para analizar la estructura poblacional de los microorganismos. La secuenciación masiva (NGS) puede aplicarse para la tipificación basada en la secuenciación 6-7 genes (MLST) o varios miles de genes (core genome ST; cgST y whole genome ST; wgST) que puede permitir en algunas especies bacterianas, como K. pneumoniae, la diferenciación de diferentes linajes dentro de un mismo clon o ST51. Las técnicas de NGS son especialmente útiles por su elevado poder discriminatorio cuando se analizan los cambios a nivel de cada nucleótido (SNP), pues en este caso se pueden identificar pequeños cambios entre cadenas de transmisión que están muy relacionadas, lo que permite conocer con gran exactitud cómo se originan los brotes y cómo se van generando nuevas cadenas de transmisión desde el caso índice53.
Informe acumulado de sensibilidad antimicrobianaEstos informes son muy relevantes para conocer las tendencias temporales de la sensibilidad a los principales antimicrobianos de uso clínico y para la selección de tratamientos empíricos adecuados. Constituyen una herramienta esencial para el trabajo de los equipos PROA y para el desarrollo de las guías de tratamiento empírico locales54. La mayoría de los centros siguen la normativa de EUCAST para su desarrollo55. Como normas generales:
- •
Los informes suelen ser anuales.
- •
Debe incluir los antimicrobianos que se detallan en los informes de resultados habituales.
- •
Normalmente se combinan las categorías S e I.
- •
Debe informar los porcentajes de los mecanismos de resistencia de interés epidemiológicos (ej. carbapenemasas, BLEE, etc.).
- •
Debe incluir exclusivamente los aislados de muestras clínicas que hayan sido validadas por un microbiólogo clínico.
- •
Deben incluir un solo aislado por paciente (el primero).
- •
Deben exponer los resultados en porcentajes, a nivel de especie bacteriana y solo cuando exista un número ≥ 30 aislados.
Es práctica habitual diferenciar los resultados de algunos servicios o unidades especiales. Las tasas de sensibilidad a los antimicrobianos en las UCI suelen ser inferiores a las del conjunto del hospital, por lo que su diferenciación es muy importante para la toma de decisiones en estas unidades56. Una de las principales limitaciones, dependiendo del tamaño de la unidad, es que a veces no es posible alcanzar el número mínimo de 30 aislados. En estos casos no se deben informar porcentajes, o si se hace se especifica el valor relativo.
Diseño de medidas de monitorizaciónExiste actualmente un debate en torno a la utilidad de los estudios sistemáticos de portadores como herramienta adicional en los programas de control de la transmisión nosocomial de BGN-MR57. La vigilancia activa de portadores de E-BLEE no ha demostrado disminuir la transmisión, aunque sí proporciona un elevado valor predictivo negativo58. Por este motivo no se recomendaría la monitorización sistemática de E-BLEE, excepto en situaciones de brote con el objetivo de dimensionarlo. Otra cuestión diferente es utilizar los resultados de los cultivos de vigilancia epidemiológica para la elección del tratamiento empírico de los pacientes inmunocomprometidos en UCI59. Con este objetivo sí que tendría sentido monitorizar tanto E-BLEE como el resto de BGN-MR60. Otra cuestión diferente es utilizar los resultados de los cultivos de vigilancia epidemiológica para la elección del tratamiento empírico, aspecto controvertido por el limitado valor de este tipo de muestras para predecir la etiología de las infecciones de pacientes de UCI59,60. En cuanto a la vigilancia de otros BGN-MR, como A. baumannii, P. aeruginosa o EPC, no se dispone de evidencias tan claras, por lo que la mayoría de los consensos continúan recomendando su monitorización en pacientes que ingresan en este tipo de unidades.
Los pacientes una vez ingresados en la unidad están expuestos a reservorios ambientales, como las superficies contaminadas y las biocapas bacterianas de los sifones61,62. Esto justifica la monitorización durante todo su ingreso, aunque no se dispone de datos sobre la periodicidad óptima para ello. También es importante, cuando se produce un brote, así como en situaciones de alta o prolongada endemicidad, los estudios de muestras ambientales pueden servir para detectar reservorios o fallos de limpieza o descontaminación. Uno de los reservorios más importantes son los sifones de los lavabos de la UCI, que han demostrado ser el foco de muchos brotes en estas unidades63. No obstante, fuera de los escenarios mencionados, no se recomienda el muestreo sistemático, ya que estos sistemas acuáticos están con frecuencia colonizados sin que se haya demostrado una repercusión significativa en la presión de colonización64. Otro aspecto importante es la monitorización de los dispositivos semicríticos, como los endoscopios65, o los dispositivos no críticos, pero próximos al paciente, como colchones antiescaras o mesas66. En el caso de que no se utilicen broncoscopios desechables, el control microbiológico de la limpieza y reprocesamiento de los broncoscopios flexibles debe estar dentro de la sistemática general del hospital de monitorización de limpieza y desinfección.
Estrategias de comunicaciónTodos los pasos comentados necesitan un buen sistema de comunicación tanto con los responsables de las UCI como con los equipos de infección nosocomial67. Ningún resultado rápido y veraz tiene valor si no hay un clínico detrás que recibe esta información de manera inmediata y toma las medidas oportunas. Actualmente, los sistemas informáticos de los laboratorios emiten resultados en tiempo real, se incluyen los informes en la historia clínica electrónica del paciente y permite establecer alertas que permitan al clínico conocer información relevante de manera inmediata. El uso de alertas en los teléfonos móviles se ha mostrado como una medida muy eficaz. En cualquier caso, la comunicación oral o presencial del microbiólogo con los clínicos de las UCI sigue siendo una herramienta muy eficaz para el traslado de información relevante y la discusión de otras medidas diagnósticas o terapéuticas a tomar. Se deben fomentar las reuniones periódicas entre clínicos expertos en enfermedades infecciosas, intensivistas y microbiólogos para discutir los casos más relevantes y para ver qué medidas diagnósticas adicionales o terapéuticas se deben tomar. También es relevante la celebración de reuniones, al menos una vez al año, donde los clínicos expongan las necesidades diagnósticas que necesitan en cada momento de los laboratorios de microbiología. Finalmente, es importante la colaboración de los especialistas en cuidados intensivos y microbiólogos en el contexto de las comisiones de infecciones nosocomiales de cada centro.
Los laboratorios de microbiología, en colaboración con los equipos PROA, deben decidir qué información suministrar y cuándo hacerlo. Esta manera de actuar adquiere especial importancia en las UCI68. La detección e información de determinantes de resistencia es controvertida, porque las agencias internacionales recomiendan que se informen solamente con fines epidemiológicos. Hoy en día existen numerosos sistemas informáticos de apoyo al diagnóstico y a los programas PROA que tiene en cuenta los datos diagnósticos, de resistencia y, a veces, de consumo de antimicrobianos que facilita la toma de decisiones69.
La comunicación entre diferentes centros es importante, especialmente si se reciben pacientes con BGN-MR de otros centros sanitarios, para poder identificar pacientes portadores de BGN-MR y así evitar la posibilidad de que se produzcan brotes.
Conflicto de interesesLos autores declaran no tener ningún conflicto de intereses.